# jrfrr-2025 : Journées du Réseau Français de la Recherche Reproductible
**Document collaboratif pour les notes pendants les présentations et les questions**
[TOC]
# Jeudi 3 avril
## 10h00-10h30 Ouverture
## 10h30-12h20 Session 1 : Reproductibilité et pratiques éditoriales
### Quentin Petitjean (INRAE Avignon) : Publier ses données et son code pour une recherche transparente – quelles sont les attentes des éditeurs et comment y répondre ?
#### Prise de conscience et évolution des pratiques en écologie
- 2000
- En écologie, entre 2000 et 2010, prise de conscience du problèmes (observationnels, computationnels, et probablement statistiques aussi) de réplicabilité/reproductibilité des études
- Reproductibilité computationnelle (code & données)
- Quelques cas de méconduite scientifique avec des données fabriquées
- 2010 - 2020 : amélioration des pratiques, plus de transparence des données et du code, dépôts en ligne et serveur de pré-print
- 2020 - ... : la communauté s'empare du sujet (principes FAIR), création de réseaux (RFRR, SORTEE)
##### Quelques problèmes classiques et édifiants
Sondage auprès de 1500 chercheurs (Baker, 2026, Nature) :
Causes de la non reproductibilité (1,500 scientists lift the lid):
- cherry-picking
- design mal adapté
- pouvoir statitiques faible
- Fraude:
- Exemples en écologie marine ([Oona Lönnstedt et Peter Eklöv](https://www.science.org/content/article/researcher-swedish-fraud-case-speaks-out-i-m-very-disappointed-my-colleague); [Danielle Dixson](https://en.wikipedia.org/wiki/Danielle_Dixson))
- Fabrication de données : détection de données répliquées, incohérence des relevés
- Exemple en écologie comportementale de [Jonathan Pruitt](https://en.wikipedia.org/wiki/Jonathan_Pruitt)
- Grosse réactivité de la communauté pour évaluer la fiabilité des données -> 19 rétractations à ce jour
- problème de l'évaluation du code et des données par les relecteurs, comment détecter les problèmes ?
Sondage de 800 chercheurs en écologie/évolution
- 50% des chercheurs ont eu recours à des pratiques questionnables
##### Pratiques problématiques
- Cherry-picking: sélectionner des données qui nous sont favorables (exclusion de données, ne pas reporter les variables/co-avariables)
- Faible puissance statistique: échantillon trop petit, pas assez de réplicats, puissance statistiques de l'ordre de 40%
- P-Hacking : ré-échantillonner jusquà obtenir P < 0.05, arrondir P = 0.051 -> P = 0.05, changer les analyses en cours de route ou changer de variable réponse (toi aussi tu peux devenir une star grâce à cette [méthode simple](https://stats.andrewheiss.com/hack-your-way/) ! )
- HARKing: présenter une hypothèse après avoir observé les résultats d'un modèle. Possible de détecter avec une méta-analyse
##### Méta analyse mésange bleue / Eucalyptus
- 2 jeu de données soumis à analyse à 173 équipes
- questions posées identiques
- mais choix des méthodes libre
- mésange bleue
- Majorité des résultats dans la même direction mais significativité variable
- Eucalyptus
- Analyses ont des conclusions différentes (même si majorité pour non significativité)
##### Avancées en science ouverte
Entre 2015 et 2019, plus de partage des données en hausse mais le code partagé progresse moins.
Besoin de réformes des pratiques éditoriales pour favoriser le partage des données et du code pour améliorer leur vérifications
##### Groupes qui se sont emparés de la question
- Mise en place de guide de bonne pratique
- 275 journaux en E/E
- 38% obligent à partager les données
- 27% obligent au partage du code
- Ouverture d'un poste de data/code editor côté journal pour évaluer les données/code soumis
Ajout du rôle de Data editor, en charge du contrôle de la qualité des données.
- 6 grandes étapes pour favoriser/s'assurer de la réutiilisation, transparence & reproductibilité compoutationelle
- Archivage des données et métadonnées (étape 1 et 2)
- contenu des données respecte les principes FAIR
- dans un format interopérable
- dépot public avec DOI et licence de réutilisation
- données doivent être citées dans l'article
- Archivage du code archivé et annoté (étape 3 et 4)
- Code archivé avec les principes FAIR (DOI, licence, )
- Documentation de la reproduction dans un README, annotations du code
- Citation du code dasn l'article
- Rééxécution du code avec les données et vérification que les résultats correspondent (reproductibilité computationnelle)
- Reproduction des calculs, tableaux et figure
##### Pour résumer
La communauté E/E s'est bien emparée du sujet mais des directives claires, obligatoires et contrôlées doivent être mises en place par les journaux
Être proactif n'est pas suffisant, les journaux doivent se réformer et participer au changement de culture
##### Questions
Si vous avez des questions vous pouvez les poser ici, je les poserai pour vous.
- Slide Case study (Structure and Readme): référencement dans l'article et backrefs ([1.3 et 1.5]). En pratique, c'est assez compliqué. Des bonnes pratiques à partager ?
- Comment on fait quand les analyses sont gourmandes en calcul (e.g., quelques jours) ?
- Des guidelines qui continuent à évoluer. On suggère que l'auteur puisse mettre un sous-échantillon de jeux de données sur lequel on puisse faire tourner les données.
- Sur la disponibilité du code dans les journaux. C'est sous quelle forme ?
- DOI en général ou lien vers des dépots institutionnels dont on espère qu'ils ont une certaine pérénité
Rq : pour assurer une certaine pérénité, la solution de la référence "fiche HAL logiciel + software heritage" est une bonne solution il me semble.
(https://www.softwareheritage.org/?lang=fr, https://hal.science/)
- Guidelines: Comment s'est passée la discussion avec les journaux qui peuvent être assez réticents ?
- Quelques journaux à l'avant-garde. C'est plutôt bien accepté car journaux avec un impact et reconnus dans la communauté. Des auteurs s'emparent aussi du sujet et sont pro-actifs.
- Dans votre introduction vous parlez de communautés en avance, vos guides lines sont-elles construites en regardant l'avancé de ces communautés ?
- Vous êtes vous inspirés des méthodes du génie logiciel ?
- Quelle licence est préconisée pour les Données ?
### Denis Bourguet (INRAE Montpellier) & Thomas Guillemaud (INRAE Sophia-Antipolis) : Peer Community In (PCI) et Peer Community In Registered Report (PCI RR), deux initiatives de science ouverte au service de la reproductibilité scientifique
Paradoxe: Quelle partie de l'étude ne doit-on pas interférer ? les résultats mais en même temps les résultats doivent être fantastiques pour la carrière, les financements, etc. Le biais de publication est monstrueux et peut atteindre 90% :pensive:.
Monde idéal : Qualité de la recherche évaluée sur la validité scientifique (question et méthode) mais pas sur les résultats.
Registered Report: découpler la publication de l'originalité des résultats pour limiter le biais de publications
- Idée formalisée par Christopher Chambers en 2013 au sein de la revue Cortex
- 3 étapes.
- Étape 1: contexte/questions/éthodologie. Après examen et review, obtention d'un *In Principle Acceptance*
- Conduite de l'étude
- Étape 2: Context/Questions/Méthodo, Results, Descussion. Peer review, vérification (données, scripts, etc.), demande de justifications
- Étape 3: Publication
Ce mécanisme tue un paquet de problèmes (biais positifs, p-hacking, harking, etc.)
Les Registered Reports sont devenus mainstream (350+ journaux, 1000+ RR).
Limites connues:
- Le temps de review du Stage 1 est un problème pour pas mal de personnes (difficultés à combiner avec logistique expérimentale et timing des contrats de doctorants & postdocs)
- Le stage 1 n'est pas toujours adapté quand il y a plusieurs stage 2 RRs (recherche programmatique)
- 2 autres problèmes mais pas eu le temps de noter
PCI: Peer Community In
- donner aux chercheurs la possibilité de reprendre la main sur les processus
- plateforme d'évaluation et de publication dans leur champ disciplinaire
- une 20aine de PCI à ce jour, disciplines/objets orientée
- dépôts des données et scripts obligatoires
- PCI indexé dans SCOPUS et Web of Science
PCI RR: pipeline commun pour évaluer des RR pour tous les PCI, dans toutes les disciplines, sans frais
- Transparence totale avec publication des recommnadations et des évaluations
- Publication dans Peer Community Journal ou dans les journaux PCI-RR friendly (stage 1 ou stage 2, avec éventuellement des APC)
Le problème des recherches programmatiques: PCI-RR règle le problème en permettant de lier plusieurs Stage 2 à un stage 1
Les recommandeurs (éditeurs) sont formés et doivent passer un examen pour s'assurer de la bonne acquisition de ce qu'est un RR :flushed:
Pour répondre au problème de la durée du stage 1, mise en place d'un scheduled review. Les auteurs envoient un snapshots (draft) en indiquant qu'ils enverront un stage 1 à telle date. Pendant ce temps là, l'éditeur identifie les reviewers qui auront 5 jours pour faire l'évaluation (mais ils sont prévenus à l'avance et s'organisent en conséquence). :flushed:
##### Questions
- Lien avec les financeurs ?
- En réflexion. Jusqu'ici, lien avec des financeurs de niche, mais discussion récente avec l'ANR pour réfléchir à une mise en commun des deux formes d'évaluation.
- Ça parait également complémentaire avec le comité de thèse pour une recherche programmatique.
- J'ai vu Nature dans la liste des journaux mais je n'ai jamais vu de RR publié dans Nature. Est-ce que ça ne tue pas le processus de recherche où il y a une part de choses non prévues, non anticipées.
- On a mit Nature pour faire bien mais ils ne jouent pas le jeu puisqu'ils peuvent rejeter les articles sur la base des résultats!
- RR traite du processus de publication mais pas du processus de recherche, il est possible de formuler de nouvelles hypothèses et d'acquérir des nouvelles données pour vérifer des nouvelles hypothèses. Il faut réfléchir beaucoup plus en avance à ce qu'on peut trouver.
La démarche normale incrémentale, hypothético-déductives est toujours possible. Ça permet principalement de distinguer prédictions et postdictions. Et ça permet de publier les résultats négatifs.
- Comment faire adhérer les autres communautés des chercheurs au PCI puisqu'actuellement c'est surtout les "sciences du vivant" qui s'y mettent ?
- Un peu vrai, mais ça percole et on a de la psycho, de l'organization studies (SHS), de l'archéologie, etc. Et la plupart des RR qu'on reçoit ne sont pas en science de la nature, mais plutôt en psycho, SHS, écoonmie. Un PCI économie est en cours de montage mais besoin de bonnes volontés.
- Les cultures et pratiques (bibliométrie, SIGAPS) expliquent aussi des dynamiques différentes d'un domaine à l'autre.
- Processus impliquant une énorme transparence, est-ce que ça ne pose pas d'autres problèmes, notamment les acteurs industriels qui ne sont pas forcément soumis aux mêmes règles.
- C'est un problème général qui dépasse notre cadre. Il faut savoir que le stage 1 peut rester privé et n'être révélé qu'au moment de la publication du stage 2.
- Sur le problème de la licence morale, c'est un concept difficile à définir.
### Sandra Guigonis (OpenEdition Marseille) : Science ouverte et reproductibilité : de quelques enjeux, pratiques et usages en sciences humaines et sociales
OpenEdition: infrastucture de recherche
- UAR CNRS, Aix-Marseille Univ., EHESS, Avignon univ., MESR, programme Investissement d'avenir
- Equipe de 70 personnes, majorité IE/IR
- initiative lancée dans les années 90 pour mettre en ligne la recherche en SHS (en Histoire historiquement)
- 1999: création d'un système de publication en accès ouvert, en texte intégral en Histoire
- 4 plateformes, basées sur des logiciels libres
- 1 million de documents (97% en accès ouvert)
- OpenEdition Journals
- [Calenda](https://calenda.org/)
- Hypothèses (carnets de recherche)
- OpenEdition Book
#### Emergence de la question des données
Enjeux autour des donneés, de la transparence, de la science ouverte plus qu'en terme de réplicabilité
2010: émergence des *Digital Humanities* (humanités numériques)
- Manifeste des humanités numériques: questionne la manière dont on produit et diffuse les savoirs
- Lier les SHS avec le numérique
Données internes au sein d'OpenEdition
- Inventaire, quelle ouverture ?
- logs serveur, indicateurs (statistiques de publications, indicateur d'activité)
- corpus
- texte intégral en XML-TEI : déjà scémantisé, avec une sémantique
- métadonnées et texte intégral moissonnables via OAI-PMH
- pas ouvert à tout le monde (politique d'embargo, ouvrages non ouverts)
- questions de recherche autour de ces corpus
- comment lier les contenus entre eux (outil Bilbo d'enrichissement des références bibliographiques)
- comment les gens s'approprient le savoir (analyse des logs)?
- Réflexion autour d'un portail OpenData
- audit sur les principes FAIR
#### Projets en science ouverte
##### I FAIR IR
Implémentation des principes FAIR dans les Infrastructure de Recherche
Association OpenEdition et Métopes
Élaboration d'un ensemble des pré-requis
- attribution de DOI à l'ensemble des documents/contenus
- licence claire sur tous les contenus diffusés sur les plateformes
- application qui permet de transformer les formats textes, permettant l'interopérabilité
- schéma XML-TEI commun aux 2 plateformes,
Où en est-on?
- Politiques de licence déployées, contenus en CC0, etc. Ça pris 3 ans et ce n'est pas terminé. C'est compliqué car on n'est pas propriétaire des contenus mis à disposition sur nos plates-formes. Il fallait donc travailler avec les éditeurs pour trouver les modalités adaptées à leur contexte (même si nous, on recommande CC-BY-SA) et qu'ils puissent prendre une décision éclairée sur la licence par défaut à appliquer à leurs contenus, plus travail juridique sur les CGU de nos plates-formes. La part de contenus en "Tous droits réservés" diminue dont petit à petit et les revues vont vers des licences de plus en plus ouvertes.
- Fait au niveau d'OpenEdition, restera à le faire au niveau de hypothese.org...
##### Autre projet: COMMONS
COnsortium of Mutualised Means for OpeN data & Services for SSH. Un Equipex+/PIA3.
- Consortium OpenEdition + HumaNum + Métopes
- Offre de services liant données et publications
- S'appuie sur les principes FAIR
- Ouverture des publications et des données et leur mise en lien
On va y retrouver :
- des objets multimédias
- des donnees quantitatives
- des complements à la publication
- des formats emergents
Usage des données en SHS (Lisa Harper, 2023):
Difficultés liées aux données
- manque de temps
- difficile à trouver
- Différence entre ceux qui partagent et ceux qui réutilisent
- quel est l'intérêt de réutilser des outils ou données mis à disposition par d'autres ?
##### Questions
- Un gros focus sur les données, mais qu'est-ce qui est fait pour le code de la recherche ?
- Actuellement, dans des articles, on ne peut pas mettre à disposition du code exécutable. Si le code est disponible quelque part, on peut y donner accès (ex: [Rubrique GeopenMod](https://journals.openedition.org/cybergeo/23412#tocto1n3) de la revue Cybergéo).
- code produit par OpenEdition en licence ouverte
- Lien avec SWH ?
- Travail en cours, dans le cadre de COMMONS et HumaNum
- Q : Un travail a-t-il mené non pas sur les datapapers mais les données liées par les revues (vous avez parlé d'annexe..)
- Oui, ça a été fait de façon systématique (peut-être dans le rapport d'activité), en analysant des liens, souvent mis à disposition sur nakala. Beaucoup de documents donc écgantillonnage pour creuser la nature des documents, évaluer l'exposition du lien, etc.
- Q2 : un des pbs n'est-il pas la difficulté à afficher des images, vidéos sur OE ? et donc pas possible d'avoir des données de cette sorte d'où peu de datapaper en SHS.
- Exposition des videos depuis l'article. Sont interesées par tout ce qui accompagne la video (metadonnées) et que la video soit accompagné d'un DOI.
- Quels arguments sont avancés pour ne pas ouvrir les métadonnées? Si métadonnées en CC0, ça veut dire plus d'attribution, donc invisibilisation de votre travail et accaparement possible.
- Non, ça, c'est tout ouvert.
- Pour la 2ème question, ce n'est pas que le fruit du travail d'OpenEdition et ça a a été discuté avec les juristes.
## 12h20-13h40 Pause déjeuner
## 13h40-15h30 Session 2 : Reproductibilité et santé humaine
### Camille Maumet (INRIA Rennes) : Variabilité des résultats en imagerie cérébrale au travers de différents pipelines d'analyse
- Une reprodutibilité faible (11% en cancéro, 36% en psycho, 44% en médecine) qui induit perte de temps et d'argent mais surtout une confiance réduite non seulement vis à vis du grand public mais aussi de la communauté scientifique
- Solutions et problematiques differentes en fontion de la discipline mais le debat implique tous
- Idéalement, on aimerait que les sésultats restent toujours vrais même s'il y a de légères différences entre données et techniques d'analyses
- Montagne de question qui se posent si ça ne se reproduit pas:
- compréhension de la méthodologie
- difference dans les données, plataforme de calcule, autres differences
- raisons computationnelles (versions etc)
- parfois certains détails ne sont pas présents dans les articles car considérés comme sans grand intérêt (le masque) mais sont importants pour reproduire les résultats
Vocabulaire :
- reproductibilité computationnelle (collègues informaticiens)
- réplicabilité large (neuroscience)
Reproductibilité computationnelle
- difficile à faire, décrire très précisément les librairies logicielles
- attendre des resultats egaux bit à bit, c'est encore difficile aujourd'hui
- Pas même données initiales : questionnement puissance statistique insuffisante
- Données fixes et on fait varier l'analyse: le point qui va nous intéresser ici (en écho à la présentation de ce matin en écologie)
- multiplicité d'analyses
- Données d'entrée : IRM
- Données en sorties: carte d'activation (où et quand, variation selon la tâche)
- Analyse en 2 blocs
- préparation des données (bruitage, correction du mouvement, alignement des référentiels, etc.)
- analyse statistiques
- Données derivées (anatomie, segmentation, etc)
- décomposable en plusieurs sous étapes
- à chaque étape, les praticiens/analystes font des choix (algorithmes, logiciels, version de logiciels, choix des paramètres, environnement logiciel)
- la famille d'analyses a plus de 10^30 combinaisons (ça reste beaucoup, même en excluant les combinaisons qui n'ont pas de sens ou sont considérées comme de mauvaises pratiques par la communauté)
- plus on a d'étapes, plus on a de compléxité
1 jeu de données d'entrée et 9 questions oui/non [Botvinik-Nezer et al., Nature, 2020]
- 70 équipes qui ont fait des analyses
- pas une équipe n'a fait les mêmes choix
- Les résultats contradictoires (équipes en desacord)ont été fréquents.
- 50/50 sur certaines hypothèses
- Cela a initié une demarche dans la communauté pour mieux comprendre la variabilité analytique.
**variabilité analytique**: variabilité observée dans les résultats quand la chaine de traitement varie
Causes de la variabilité analytique (essential pour identifer ce qui est intéressant)
- incertitudes numériques ou liées au logiciel ($\ne$ OS)
- variabilité liée aux données (jeu de données pas adaptées à la question posée)
- implémentations différentes
- résultats instables
- violation des hypothèses
- variabilité d'intérêt (ajout de co-variables)
Balance pour comprendre quelle part de cette variabilité analytique est interesante et peu aporter à la recherche et de quel part on voudrais pouvoir controler
Constuire un jeu de données pour explorer l'espace des résultats (Thèse d'Elodie Germani sous la direction d'Élisa Fromont)
- 24 types d'analyse en faisant varier le logiciel, un paramètre de pré-traitement, 2 paramètres statistiques
- 1 jeu de données ouvert (1000 participants, ouvert) HCP Young Adult
- 4 clusters qui se comportent de manière similaires et pour chaque cluster, choix d'un représentant
- Hélas, d'une tâche (main droite, main gauche) à l'autre, les clusters sont assez différents. :frowning:
NARPS open pipelines
- vu précédemment (70 équipes, 9 questions)
- fournis carte et analyse textuel (COBIDAS)
- pas de code
- brainhack.org: projet collaboratif pour reproduire ces pipelines. Venez contribuer!
Quelles solutions ?
- proposer des méta-analyses
- méta-analyse traditionnelle : indépendance des données d'entrées
- **analyse multiverse**: même jeu de données d'entrée. Proposer des méta-analyses qui fonctionnent sur les mêmes données (prendre en compte la correlation)
Perspectives: l'status quo est resultats avec 1 unique analyse. Dans le futur l'ideal sera de:
- faire varier plus de paramètres
- détermination des principaux axes de variabilité
- stabilité de certains axes
- 3 axes de travail
- Modéliser l'espace ds analyses
- tracer la provenance
- Explorer l'espace. pas eu le temps de noter (completer avec les diapos)
#### Questions
- Problème de la vérité terrain en imagerie cérébrale: pas de façon facile de dire ce pipeline fait mieux que celui-là
- méthode de proxy: par exemple prédire l'âge à partir des images, pose plein de questions
- Consensus:
- Est-il possible d'avoir un niveau de confiance dans certaines analyses ?
- sur les paramétrages
- Estimation du movement, le signal qui correle qu mouvenent n'est pas très interesant parce qu'elle est probablement due au mouvement.
- Les praticiens ont des contrainte de temps. Comment faire ?
- Analyses 20min par pipeline et par participant, s'il faut repeter c'est trop. Il faut arriver à reduire le nombre de pipelines
- Vers une méthodologie de type analyse de sensibilité des différents paramètres ?
- Définir les metriques de sortie. Elle fait des correlations mais peut etre pas ideale
- Utiliser l'analyse de sensibilite pour reduire les pipelines. Pour l'instant ils n'en sont pas là parce que il faut que ça s'applique à tous les données.
- Pour la reproductibilité, en vous écoutant, j'ai l'impression que l'on prend le problème à l'envers en partant des résultats au lieu de partir du besoin. Utilisez-vous des méthodes d'analyses du cahier des charges du génie logiciel pour gérer la reproductibilité ?
### Tristan Glatard (Centre for Addiction and Mental Health, Toronto) : Reproductibilité des analyses IRM pour la maladie de Parkinson
Lien Zoom pour la présentation :
https://cnrs.zoom.us/j/98703507964?pwd=01YIv1MIBAbBOnwUX1UbgyKqXxiOyT.1
Petite remarque concernant le zoom du CNRS: il n'est pas possible de se connecter via le client web, c'est une option dans la configuration de la réunion ("chiffrement standard" de mémoire), tout le monde n'a pas le client lourd zoom sur son ordi : merci pour la remarques, dsl je ne savais pas. On fera mieux pour la visio de demain du coup.
Par contre, la bonne nouvelle, c'est en direct sur Canal-U aussi (méli-mélo d'ordi qui fonctionnent finalement)
Question principale: est-ce que la (non-)reproductibilité des traitements a un impact sur l'analyse clinique ?
IRMs utilisés pour comprendre Parkinson
- IRM anatomique, messures de volume, taille, etc
- IRM de diffusion
- IRM fonctionnelle
Maladie de Parkinson méconnue. Beaucoup d'articles utilisnt l'IRM mais cette dernière n'est pas utilisée cliniquement alors qu'elle est peu invasive. Cela s'explique peut-être par les doutes en terme de reproductibilité ?
Usage des IRM:
- diagnostique : syndrôme parkinsonien (syndrômes identiques mais ne sont pas la maladie) vs. maladie de Parkinson.
- prédiction de la progression de la maladie (difficile à prévoir). Interet dans la discipline a identifier si des trajectoires differentes de progression peuvent étre sensibles à differents traitements.
-la taille des effets est faible par rapport à la variabilité des mesures avec les méthodes actuelles. Cela peut constituer une source de problèmes de reproductibilité dans la communauté.
- Il y a quand même des résultats robustes avec la méthodologie, comme l'effet de l'âge, qui est mesurable de manière reproductible avec les pipelines d'analyse
#### Rôle de l'environnement d'exécution
- Variabilité importante mesurable liée à l'environnement logiciel
- plusieurs segmentations correctes mais avec des variations
- analyse fonctionnelle sensible à l'environnement
- facteurs d'enclenchement
- mise à jour logiciel
- mise à jour matériel (CPU vs GPU)
- variabilité mesurée avec l'arithmétique de Monte-Carlo
Variabilité numérique
- dépend des données
- impacte les populations saines et cliniques
- impacte tous les types d'IRM
- dépend des paramètres d'analyse
- impacte outils Deep learning mais peu de variation inférence réseaux de neurones convolutionnels
- Variabilité numérique impactée de manière similaire selon l'environnement
- utilisation de containers ne masque pas la variabilité numérique dûe au matériel
- modèle de deep learning varient peu à l'inférence mais celle de l'entrainement peu/pas connue
- Opportunités associés à la variabilité
- augmenter les données pour l'apprentissage automatique => meilleure généralisation des modèles
- concevoir les tests logiciels (parce que pas de vérité terrain)
- perte de précision volontaire => temps de calculs plus rapides
- calcul sur des zones images qui n'apportent rien (contour, certaines zone sdu cerveaux)
#### Impact pour la recherche clinique
Impact important et mesurable dans les 2 cas
##### Variabilité logicielle
- Comparaison de résultats de 300 sujets en faisant varier les versions de Freesurfer
- différences statistiquement significatives entre les mesures
- mesure du volume: la variabilité logicielle approche la variabilité anatomique dans plusieurs cas
- correlation avec les variables cliniqiues (sévérité de la maladie): résultats différents
Recommandations
- ne pas chager deversion pendant les études
- utiliser la dernière version
##### Variabilité numérique
Dans bien des cas, les environnements d'exécution vont avoir des résultats cliniques contradictoires
#### Que faire ?
Réfléchir aux paradigmes utilisés
- plusieurs niveaux (pipeline de pré-traitement, statistiques, généralisation)
- Challenges / compétitions de type kaggle et trouver la / les meilleures solution (type IA) évaluées avec métriques reproductibles
- évaluation systématique des résultats: niveau de preuve beaucoup plus important
- Brain Health Data Challenge Platform
- consortium
- résultats plus reproductibles, plus robustes
#### Questions
- Si nous avions la variabilité numérique mise à jour suite au changement de logiciel ou matériel, alors serait-il encore utile de s'en préoccuper ?
- Question pas hyper claire... Ça veut dire quoi mise à jour ?
- Mise en place de challenge : quel financement ? Quel gain pour les chercheurs ?
- Problèmes de variabilité de l'IRM liés aux populations (sexe, cohortes, etc.), à l'acquisistion, aux imageurs, aux antennes utilisées, etc.
- Différentes façons d'approcher ça. Projet Enigma avec problème de biais multi-centrique. D'un point de vue ML, on pourrait vouloir entraîner des modèles ayant vu ces différents biais.
### Florian Naudet (CHU Rennes) : Intégrité et reproductibilité en recherche thérapeutique
Recherche en thérapeutique: efficacité des thérapeutiques ?
- taille d'effet parfois réduite
- problématique de la sécurité des médicaments
- Production à l'échelle industrielle -> scandales sanitaires à l'échelle industrielle également
- Président Kennedy signe un décret en 1962 pour rendre obligatoire l'évaluation de l'efficacité des médicamments par essais randomisés controlés
- 1959: Louis Lasagna: étude randomisée en double aveugle sur l'impact de la Thalydomide (mais scandale non évité)
- [Free lunch index](https://hal.science/hal-04286546): somme de nourriture offerte par les laboratoires aux cliniciens, assez corellée au h-index :stuck_out_tongue_winking_eye:
- Pyramide des preuves: cas cliniques -> études observationnelles -> essais randomisés -> méta-analyses
Et pourtant: [A survey of biomedical journals to detect editorial bias and nepotistic behavior](https://journals.plos.org/plosbiology/article?id=10.1371/journal.pbio.3001133)
Comment faire la différence entre les études robustes et les autres (projet OSIRIS Delphi)?
- enregistrement à priori non consensuel
Cube:
- flexibilité des méthodes (faire un varier une seule variable)
- flexibilité des hypothèses de recherche
- analyse primaire, analyse secondaire, réutilisation de données
- flexibilité de mesures
Méta-analyse en réseau (20 anti-dépresseurs, tous les réseaux possibles): 8 million de méta-analyse
- comparer les médicaments pour trouver le meilleur
- quand industriel finance l'analyse, son produit est parmi les meilleurs
Biais de publication:
- certaines études ne sont pas publiées
[Selective publication of antidepressant trials and its influence on apparent efficacy](https://pubmed.ncbi.nlm.nih.gov/18199864/)
- certaines études négatives sont publiées comme positive
Revue post-publication
- pas efficace
Partage de données
- pré-enregistrement prospectice
- report des résultats (toutes les études ne sont pas complétées, et toutes les études complétées ne sont pas publiées)
Politique de partage de données
- financeurs industriels (100%)
- financeurs non industriels
- 40% environ
Données individuelles de patients => sensibles, très compliquées
Il faut que les structures et infrastructures soient prétes à le faire et la formation pour le faire
Réseau de doctorants pour former au partage de données et à la réutilisation
Haute Autorité de Santé: intéressé par les registered reports
Lancet a pratique les RR entre 1997 et 2015
GRIOS: Global Research Initiative on Open Science
#### Questions
## 16h00-18h00 Session 3 : Reproductibilité et recherche préclinique
### Christophe Soulage (Université Lyon 1) : Crise de la reproductibilité en recherche préclinique : état des lieux et solutions
Recherche préclinique -> pas chez l'homme
Comment utiliser les modèles animaux pour trouver de nouvelles cibles thérapeutiques ?
Taux d'attrition des molécules de 99.99 % (1/10,000 molécules passe à l'homologation)
Cible : mieux comprendre la maladie et développer un médicament qui aurait un impact sur celle-ci. Grosse lecture de la litterature scientifique pour identifier les cibles.
Réplication des travaux par les industriels pour diminuer les risques
- parfois ces travaux sont publiés
- méthodoloogie souvent plus rigoureuse qu'en recherche académique
Sur 685 études, 57% ont pu être reproduites (étude de 2015)
Pour Bayer, 86% des études reproduites dans des conditions différentes (modèles naimaux différents) mais résultats similaires => hétérogénéité améliore la robustesse
Recommandations AMGEN : répéter (même labo), répliquer (autre labo), faire varier les modèles animaux
Réplication d'une étude par Novo Nordisk
- souris diébétique) : aucun effet mesuré lors de la réplication (dans plusieurs centres)
- GABA : pas d'augmentation des cellules pancréatiques
- manque de robustesse des travaux académiques (protocoles pas assez rigoureux, réactifs de mauvaise quelité, pas assez de procédures)
~50 % des études non répliqués -> 28 milliards € de recherche inutiles
[Loi d'EROOM](https://en.wikipedia.org/wiki/Eroom%27s_law): le coût pour développer une nouvelle molécule sur le marché double tous les 9 ans
- dûe au manque de force statisitque (?)
- Comme d'hab: Harking, p-hacking, etc. comme listé dans [A manifesto for reproducible science]([https://www.nature.com/articles/s41562-016-0021).
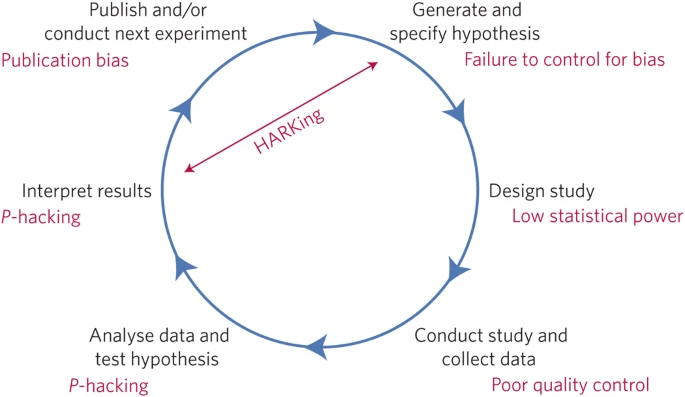
6 péchés capitaux
- mauvais design expérimental (pas de randomisation)
- faible puissance statistique (pas de calcul a priori du nombre de sujets nécessaire)
- bidouillages statistiques (p-hacking)
- changement a priori des critères de jugement (HARKing)
- En expérimentation animale, pas d'obligation d'enregistrement des expériences avant les années 2000. Après 2000: Clinicaltrials.gov.
- biais de publication
- 2007 moins de 15% des articles en biologie publient des résultats négatifs
- Excès de standardisation (souches extrêmement pures et contrôlées: amélioration de la reproductibilité/validité interne, i.e., résultats valables sur une souche de souris donnée/dans un laboratoire/etc., autrement dit pseudo-réplication vs. replication, les souris de laboratoires n'étant plus représentatives)
- plasticité phénotypique : d'une souche de souris à une autre on peut avoir des résultats différents
- résultats ne s'appliquent que dans des conditions très particulières
- recherche de résultats plus généralisable : gagner en validité externe et en transposition
- inclure des animaux de plusuieurs sexes
- téudes multi-centriques
- plusieurs cohortes d'animaux
- différentes sources animales
Que faire ?
- science ouverte
- pré-registration avec PCI (pas encore reconnue par les institutions)
- Plus de crédits pour les études de réplications
- Développement de preclinical Randomized Controlled Trial

#### Questions
- Essais sur 2 ou 3 primates (déjà hyper coûteux mais très faible puissance statistiques) : validité ? pas assez d'animaux, cohortes trop petites pour les essais, mais problèmes éthiques évidents, ainsi que d'approvisionnement
- De plus le cycle jour-nuit, la température, l'humeur et l'expérience de l'expérimentateur ont un impact sur le résultat des expériences (au moins pour du comportemental).
- Comme disait Ioannidis, la plupart de nos résultats publiés sont faux car on mesure de la fluctuation expérimentale.
### Marlène Wiart (CNRS Lyon) : Des 3R aux 6R, ou comment la mobilisation de la communauté AVC a révolutionné la recherche préclinique
Si résultats positifs en tre plusieurs centres, plus de chance que ça marche en clinique
AVC: première cause de handicap chez l'adulte, deuxième cause de décès
Sur les patients recanalisés (arthère débouchée mécaniquement) , 50% gardent des séquelles
Neuroprotection: protéger le cerveau des effets secondaires de la reperfusion
- années 90:
- avancées IRM et thrombolise (traitement d'urgence de l'AVC)
- 1996: FDA approuve le traitement par thrombolise
- 1999: conférene STAIR (recommandations pour l'évaluation de molécules neuroprotectives)
- sélection d el'animal
- modèles d'AVC
- 2004: initiative CAMARADES
- 1,026 treatments in Acute Stroke (et aucun ne marche :shrug:)
- graphe nombre critères STAIRS appliqués x effet neuroportecteur
- mesure du volume impacté
- Une des étude avec gros niveau de qualité met en avant l'efficacité de la molécule NXY-059
- 2007: SAINT II : molécule NXY-059 très bien classée précédemennt mais pourtant pas efficace
- analyse des études qui ont évaluées la molécule
- peu de randomization
- souris rarement hypertendues hors AVC plus fréquent chez les personnes hypertendues
- 2009: STAIR IV
- nouvelles recommandations
- importance du reporting des conflit d'intérêt
- ajouter des comorbidités
- confirmer dans plusieurs labos
- utiliser des modèles plus proches de l'humain
- 2010: ARRIVE
- 2012: RIGOR
- 2013: projet PLUTIPART: recherche multicentrique (315 animaux)
- 2017: IMPROVE
Comparaison entre 24 animaux dans un labo et 24 animaux dans 3 labos (8 par labo). Souris souvent de type male, sans co-morbidités, etc.
- si réplicat dans 1 seul labo, plus de variance qu'en 3 autres labos qui ont des conditions différentes => effet bénéfique du multi-centrique
- Encore une fois, on est sur pseudo-replication vs. replication au niveau du plan d'expérience
-
2018: arrivée de la thromboloectomie (débouchage mécanique)
Initiative SPAN
- 6 laboratoires avec un centre coordinateur
- 6 traitements en parallèle
- cohorte 710 animaux mixte male/femelle, souris jeunes/agées
- publication 2025 : effet positif de l'acide urique comme neuroprotecteur => essai clinique ?
#### Questions
- Passage souris -> humain direct ?
- phase 0 : interaction molécule cible
- souvent passage rongeur jeune vers une personne agée
- toxicité : 1 modèle rongeur et 1 modèle non rongeur
- quand le critère principal ne marche pas, analyse exploratoire par sous-groupe pour essayer de comprendre
- question de bien valider en pré-clinique avant de passer en clinique
- environnement très propre et très contrôlé, souris de laboratoire très différent d'une souris sauvage
- changement des pratiques pour utiliser des animaux qui soient pas dans des cages mais dans des environnements plus "naturels"
- C'est quoi le principe du 6R ?
- 3R = Réduire, remplacer, rafiner
- Bien-être animal non éthique si résultats non scientifiques
- 6R: 3R précédents, plus:
- reporting
- registration
- robustness
- faire en sorte que tous les animaux utilisés dans l'expérimentation animale aient une valeur ajoutée
### Laura Barrot (INSERM Lyon) : Aide au design des projets précliniques pour une bonne science et une bonne conscience
- Coût financier et éthique de la recherche pré-clinique
- Éthique du patient lié à molécules plus ou moins efficace
- Mais aussi éthique animale car le regard de la société a évolué
- A-t-on le droit d'avoir recours aux animaux en recher ? Non, on demande le droit à un comité d'éthique
Les attentes sociétales actuelles tendent à arrêter l'expérimentation animale
INSERM programme LORIER : construire une culture de recherche éthique et responsable. INSERM également à l'origine du GIS FC3R.
Vie d'un projet c'est long, 12 étapes de l'hypothèse à la valorisation. Longue préparation, avec des biais à toutes les étapes
- guideline PREPARE: checklist
- guidelines ARRIVE: 10 items à faire paraître sur les publications (plus 11 recommandés). Pas très vérifié
Pré-enregistrement pré-clinique
- 2 plateformes possibles (DOI, embargo de 1 à 5 ans)
- oblige à une planification rigoureuse
Facteurs de nuisance vs facteurs d'intérêts: notion souvent un peu vague chez ceux qui soumettent les études aux comité d'éthique
- facteur d'intérêt : qce qu'on veut mesurer (ça veut dire qu'ils ne se sont pas trop posé la question de ce qu'ils voulaient mesurer, donc les estimations de tailles d'effectifs risquent d'être complètement foireux)
- facteur de nuisance: tout ce qui peut faire varier les résultats (temporalité, expérimentateurs différents, hébergement différent)
4 piliers du design expériemental
- randomisation
- minimise les biais de sélection
- déterminer les effectifs avec un calcul de puissance
- réduire les variabilités
- travail en aveugle
- effet sur l'expérimentation
- lors de l'idnetification des animaux
- lors des mesures
- lors de l'analyse des données
- effet du sexe
- facteur de nuisance ? si oui, qu'est-ce qu'on fait ?
- est-ce que c'est pris en compte en clinique ? Quelles sont les conséquences ?
- évolution du nombre d'études avec des sujets mâles et femelles
- puissance statistique
- puissance moyenne de 20-30%
- règles fixées avant
- des sociétés peuvent être sollicités pour un accompagnement sur la phase de conception de l'étude et celle d'analyse des résultats
- [EDA: Experimental Design Assistant](https://eda.nc3rs.org.uk/) (outil créé par le NC3R avec un réseau de formateurs d'une 20aine de formateurs sur la France)
- permet d'apprendre
- permet de ne rien oublier
- diagramme pour les partenaires, demande de financement, comités d'éthique
- outil critique qui soulève des problèmes et fournit des conseils
- fiche complète du projet
- outil recommandé par le comité d'éthique
#### QUestions
- pour éviter de gaspiller, ouvrir ces données c'est implicite ou pas ? ça ne fait pas partie des recommandations ?
- accessibilité des données c'est important
- guideline ARRIVE insiste sur l'exhausitivé mais ne se rappelle pas si l'open source est évoqué
- J'ai uniquement traité la partie design mais c'est bien dans la partie valorisation.
- J'ai l'impression qu'il y a deux mouvements entre clinique et pré-clinique, avec l'un où on essaie de randomiser au maximum pour augmenter la robsutesse, et l'autre où on stratifie pour augmenter la reproductibilité interne
- en pré-clinique, ils essaient de calquer la clinique et essayer de s'améliorer. Regard sociétal super important parfois plus qu'en recherche clinique.
- La population générale ne croit plus en la recherche, donc besoin de transparence, en particulier dans le contexte des tensions avec les anti-spécistes.
- si on demande des données, si elles ne sont pas préparées c'est juste l'enfer, alors que fourir un DOI c'est tellement plus simple
- même pour soi-même si on doit revenir sur les données 5 ans après
- En fait, qu'on soit en agro, en écotox, en ..., on a toutes et tous les mêmes problématiques et en terme de conception d'expérience on est super nuls, non ? Comment ça se fait qu'on soit si mal formés pour des trucs de base ?
- "En biologie, on est nullissime en tant que chercheur", comment cela se fait-il ? La population générale nous prenne pour des marioles. Pourquoi ce n'est pas enseigné à la fac
- pas tant lié à la formation qu'à ce qui se passe dans les labos, les doctorants sont livrés à eux-même et doivent savoir tout faire. Le doctorant sera juste un peu mauvais partout et prendra l'habitude de travailler comme ça. On ne peut pas attendre des doctorants qu'ils sachent tout faire...
- Il faut réfléchir à un niveau systémique (l'évaluation ayant un impact négatif, induisant de mauvaises pratiques, et un comportement individualiste) plus qu'au niveau des individus. Comment réformer le système ? PCI va dans le bon sens par exemple.
- Dans les universités, il y a des formations pluri-disciplinaires, repro hackathon, on va dans le bon sens mais pas vite.
- En France, on a un système de système de recherche très centralisé donc on devrait être très bon pour travailler en collectif.
- Beaucoup de pratique en science ouverte qui font peur parce que ça fait plus de travail. Essayer de faire une bonne pratique une étude à la fois, à l'étude suivante on a appris.
- DAns les années 2000, 4 attentas à Lyon par les activistes (ouverture des cages, etc.). On s'est cachés et ce secret a alimenté le discours des antispécistes. On s'est tirés une balle dans le pied, il faut être le plus transparent possible, expliquer les enjeux, expliquer que ça fait suite à plein d'expériences in siloco, expliquer enquoi l'expérience animale a apporté des connaissances, etc. Il faut ouvrir les portes des labos.
# Vendredi 4 avril
## 9h00-10h15 Session 4 : Reproductibilité sur les temps et les espaces lointains
### Aurore Val (CNRS Aix Marseille Université) : Un long chemin vers l’Open Science en archéologie préhistorique
Aurore Val est archéo-zoologue et pré-historienne. Un point de vue subjectif.
- Intérêt pour l'inseraction entre les animaux et les hominmes, presque exclusivement en afrique Australe. 1/3 de temps sur le terrain à collecter des données primaires, 1/3 en labo à traiter les données, 1/3 à les exploiter et publier
- 3 grandes catégories de données:
- Contexte: le terrain
- Objects (outils en pierre, perles, ossements, etc.)
- Échantillons (ADN ancien, rédsidus isotopes, etc.)
- Objectif de l'archéologie = reconstuire les comportements humains. Un discours qui se base uniquement sur ce qui est découvert dans les fouilles (pas de textes), ce qui laisse nécessairement part àl 'interprétation.'.
- Scénario théorique idéal = Pompei, une société à un moment T préservé dans une couche géologique.
- En préhistoire: on imagine donc un groupe préhistorique qui habite quelque part, laisse des traces, quitte les lieux, des sédiments se déposent, un autre groupe d'humains s'installe, etc. Du coup, des traces dans des strates mais en pratique, ça n'est jamais comme ça.
- Exemple de Millie's camp où des archéologue ont demandé à un indien de venir confirmer les interprétations et gros biais.
- 2-me biais (post-dépositionnel: terrier/bioturbation, gel-dégel/solifluxion, érosion, diagénèse, etc.): il est très difficile de savoir à quelle couche appartient chaque objet. Même avec des couches horizontales parfaites, il est très difficile de savoir à combien de groupes cela correspond (1 groupe puis 10 ans ou 3 groupes en continu).
- Fouilles = destruction du contexte. On ne sait pas si on a affaire à des inumations/sépultures ou si des individus sont juste morts dans des abris. Ces corps ont souvent été découverts par des hommes d'église, avec leurs propres croyances, il y a longtemps et on a des photos de mauvaises qualité avec très peu d'information sur le contexte.
- Exxmple du site de la Ferrassie où les lieux ont tout de suite été interprétés comme des sépultures alors que ça n'a a priori pas été le cas (pas de fosse creusée ?).
- Sur le terrain, beaucoup de notes écrites à la main (carnets de fouilles) avec des dessins, croquis, coupes tratigraphiques, des explications dans une une ou plusieurs langues.
- Tout ce qu'on fouille est détruit donc la reproductibilité ne peut pas se poser de la même façon que dans d'autres disciplines. En parallèle de ce qui est détruit, tout un tas de données sont créées: la couche/strate est une unité de données créée par l'archéologue de terrain basé sur les changements de texture, de couleurs, etc. D'où l'importance d'être allé sur le terrain pour comprendre.
- On fouille très doucement et on prend beaucoup de notes, des photos et on utilise des outils pour enregistrer précisément la position d'autant d'objets que possible (station totale). Plein de questions: tout est-il tamisé ? quelle taille de tamis ? etc. Des données de terrain pas toujours mentionnées dans les articles et des fouilleurs plus ou moins précautionneux.
- Dans notre discipline, les gens se spécialisent très tôt (isotope, ADN ancien, protéines anciennes, résidus organiqus, etc.) donc on n'a pas toujours toutes les compétences au moment de la fouille. Du coup:
- Importance des données terrain
- Reproductiblité compliquée car destruction
- L'échantillonnage ne peut être géré comme en bio (influence du contexte archéologique, de ce qu'on trouve, du temps, du financement)
- Anecdote d'un chercheur qui a gardé précieusement un squelette humain sans le partager avec les autres pendant 20 ans.
- Mesures:
- Beaucoup de mesures (paléontologue), de recherche de trace/absence (stries sur les dents pour carnivores),
- Stockage et partage des données:
- En France et à l'étranger, on est tenu de faire un rapport de fouille mais consignes pas très strictes donc plus ou moins d'infos et très hétérogène. Les données brutes ne sont pas toujours partagées et c'est assez dépendant des compétences de l'équipe qui a conduit les fouilles.
- Publication des données: des pratiques anciennes qui n'ont pas énormément évolué. Un gros tableau interprétatif avec des informations quantitatives et un nombre minimum d'individu basé sur une interprétation taxonmique des restes (sans forcément exploiter le fait que les restes peuvent venir de différents individus). C'est totalement irreproductible et il n'y a aucun moyen de savoir d'où viennent les différences. Le fait que la base de données complète soit attaché à l'article est rarissime.
- Publication uniquemnet des résultats positifs, et il faut une belle hitoire. Du coup, straw-man argument où une hypothèse est gonflée un peu artificiellement. Exemple de Innovative Homo sapiens behaviours 105,000 years ago in a wetter kalahari. Fragment de quartz et d'œufs d'autruche avec rien d'original mais démontant une hypothèse dont tout le monde sait qu'elle est fausse (homo sapiens is as homo sapiens was).
- Donc pratiques traditionnelles = un PDF, avec ou sans Supplementary Online Material, et les jeunes ont besoin de publier dans les grands journaux pour avoir un poste et des financements... :shrug:
Initiatives Open Science, existantes mais pas encore très répandues en archéologie: HAL, revues en Open Access, revues
- Pratiques Open Science: HAL mais un peu pénible, OpenEdition, des répertoires de donnée assez peu utilisées. Il y a un PCI in Archaeology mais y'a pas grand chose sur la préhistoire. La plupart des préhistoriens reviewers n'ont jamais soumis un article. Probablement encore un peu de défiance/interrogation vis à vis de ces journaux nouveaux.
- Un [bel example de bonnes pratiques](https://www.oldstoneage.com) (qui a un informaticien passionné) avec un site ne open access qui regroupe toutes les données collectées par cette équipe américaine dans un site en dordogne. On est un peu limité par des moyens de compétences et de temps et on n'inclue pas assez ces aspects.gestion et données dans nos demandes de financement.
#### Questions
- Mettre les données en accès ouvert (avec des coordonnées GPS) peut conduire à un pillage des sites ?
- Oui, mais en même temps, plus c'est secret, plus ça attire l'attention. En partageant l'information avec les locaux, on rend le pilleur *persona non grata*.
- COARA, l'Europe et l'ANR imposent l'ouverture.
- Tout à fait, en Europe, les pratiques s'améliorent et vont dans la bonne direction mais il y a un décalage des pratiques avec les anglo-saxons, l'afrique du sud, etc. Ils ne sont pas soumis aux mêmes règles et n'ont pas accès aux mêmes financemnt.
- Interaction avec les géologues sur le terrain pour la stratigraphie ?
- Bien sûr, on a des géo-archéologues et géologues avec nous sur le terrain. On n'est plus jamais tout seul sur le terrain. On passe beaucoup plus de temps à discuter de la stratigraphie qu'à fouiller.
- Vous avez parlé de oldstoneage. Vous connaissez archeoviz ?
- Oui. Il y a tout un pan de la communauté qui est un peu effrayé par l'informatique alors que ce sont de supers outils.
### Françoise Genova (CNRS Strasbourg) : Étude de cas : Comment la reproductibilité est assurée dans le domaine de l'astrophysique ?
- Reproductibilité: un concept peu évoqué en astro. La **confiance** est essentielle et on va voir comment la communauté astro a construit cette confiance.
- Des infrastructures (satellites et observatoirs) mais aussi les données
- archives observatoires
- Un effort communautaire bien structuré
- Importance de croiser les données collectées sur les différents objets pour comprendre leur structure et les phénomènes à l'œuvre.
- On obtient du temps d'observation via des appels compétitifs. Données à disposition en général après une période d'embargo de 6 mois à un an. Pour ça, il faut que les pipeline de traitement des données soient au top et maintenus au long de la vie des observatoires. La qualité des données et des méta-données est assuré par les observatoires mais du coup, on a a confiance. Et ça marche parce qu'on a des standards partagés (FITS). Un standard de référencement (identifiant) des publications mis en place dès 89, bien avant le DOI. Beaucoup de travaux de standardisation qui ont inspiré FAIR.
- Ça a marché car on n'a jamais demandé aux gens qui produisaient les données de changer leur système, mais juste de les mettre à disposition avec les standards de l'International Virtual Observatory Alliance.
- Conclusion: les données astronomiques sont FAIR et largement réutilisées par celle-sci dans osn travail de recerhche. Construire la confiance (production, curation, gestion, archivage, publication) dans les données est essentiel.
- Le pilotage par les besoins cientifiques est essentiel. Les aspects sociologiques sont souvent plus importants que les aspects techniques. Des systèmes de gouvernance divers. Ils dépendent de la discipline, de la culture de celle-ci, etc.
- Un contexte qui évolue en permanence (politique, scientifique, technique).
#### Questions
- FITS: adoption ?
- Défini par les radio-astronomes. C'est parti d'un besoin et c'était à l'époque où on s'est affranchi des plaques photos. Ce format est toujours utilisé, même si des formats étendus sont apparus et qu'il y a des discussions sur de nouveaux formats.
- Vous indiquez que les journaux mettent des données à disposition. Quelle pérénité
- On les conserve au CDS. Les données peuvent être gardées par les journées mais on a un entrepot de données à côté.
- Je travaille sur EUCLID. Cette noton de FAIR n'est-elle pas liée aux budgets et aux coût des instruments.
- Oui, bien sûr, le coût des instruments et leur envergure justifie la nécessité de partager les données à l'international.
- FITS = format commun, l'IVOA = aller plus loin avec le Web et c'était bien avant que FAIR soit mis en place.
## 10h15-10h45 Pause-café
## 10h45-12h30 Session 5 : Reproductibilité sur le monde qui nous entoure
### Raphaël Royauté (INRAE Versailles) SORTEE : Une société pour la science ouverte en écologie et biologie évolutive
- Une culture des études sur du long terme (dynamiques de populations, pressions huamines, réseaux de suivi de sites), de la méta-science (méta-analyse, avec modélisation, etc.), mais dans lequel il y a aussi eu des scandales et des fraudes scientifiques (micro-plastiques, acidification des océans, SpiderGate)
- [Avalanche of spider-paper retractions shakes behavioural-ecology community](https://www.nature.com/articles/d41586-020-00287-y) (article pas en open access :rage:).
- Les araignées sociales, c'est un phénomène curieux, qui a apparu et disparu plusieurs fois. [Jonathan Pruit](https://en.wikipedia.org/wiki/Jonathan_Pruitt), spécialiste, parlait de personnalité animale, et de systèmes de castes.
- Les équipes travaillaient sur des sites différents et certaines équipes observaient des patrons observationnels de comportement très différents.
- Kate Laskowski contactée car duplication de données repérées. 3 individus venant de colonies différents ayant exactemnet les mêmes attributs de captures au centième de seconde près.
- Jonathan Pruit proposait les données aux chercheurs et chercheuses mais il copiait-collait avec des patrons de duplications.
- 19 articles retractés, ça a pris 5 ans pour certains (quand les avocats s'en sont mellés, ça a mis un gros frein). Finalement pas de patrons de castes liés à des personnalités.
- Tout ceci n'a été possible que parce que données ouvertes. Conservation des scripts et des données essentielle. Les éditeurs ont pris le problème à bras le corpts et ont fait travailler la communauté dessus. Les jeunes chercheurs (en général lanceurs d'alerte) sont les plus vulnérables dans ces situations (les publications des doctorants de Jonathan ont été passées au crible)
- SORTEE = société créée en 2019, donc antérieure à ce scandale
- 500+ membres, 50 pays, 50% de jeunes chercheurs, 3 des derniers présidents = postdoc
- 50% des memebres en écologie comportementale ou en biologie évolutive mais c'est historique
- Un comité Diversité et Inclusion.
- Une conf annuelle avec un programme en distanciel de 72h en continu! Bon, finalement 24h, c'est plus raisonnable. Une conférence sans pléniaire/track satellite mais plutôt des hackathons, des [Unconference](https://unconference.historikerinnen.ch/fr/quest-ce-quune-unconference/) avec plus d'échange que d'écoute. 2 super exposés:
- [Plenary Richard McElreach, Science is Licke a Chicken Coop](https://www.youtube.com/watch?v=d8LqFO1dk-w)
- [C. Thi Nguyen: Transparency is Surveillance](https://www.youtube.com/watch?v=8JEzXL5OXPI) (philo)
Organisation également de rencontres en marge des conférences.
### Sylvie Joussaume (CNRS, Laboratoire des Sciences du Climat et de l'Environnement) Reproductibilité dans le cas de la modélisation du climat
- Couplage de modèles terre, côte/forêts/végétations, océan, glace de mer, atmosphère, activité humaine, ...
- Simulations internationales de références (depuis 1979). La communauté défini les expériences de références
- Capables d'expliquer les observations seulement lorsque les facteurs humains sont ajoutées aux modèles
- Différents scénarios d'émissions de GES -> différentes projections climatiques
- CMIP 6 = 48 groups, 120 modèles, 312 expériences, $\approx$ 15 000 utilisateur·ices, 20 Po de données, 14M datasets. 14To de données échangées par mois
- Données FAIR avec vocabulaire commun
- Workflow:
- Des CI Atmosphère Océans (essentielle pour la météo mais moins critique pour nous), des conditiosn de forçage (dégagement CO2, particules, etc.)
- Des codes de simulation
- Dérivation sur des temps longs
- Exécution sur GENCI ou autre
- Données de sorties (vent, température, etc.), métadonnées (paramètres simulation, versions des codes, etc.)
- Passage en full open progressif
- **Traçabilité et authenticité des données**: Logiciel commun npour gérer les versions de données depuis CMIP5, service de correction des données errata depuis CMIP6, service de DOI sur les données (par modèle/expérience) et identificateurs par fichiers
- Archivage long terme par le DKRZ
- Logiciels d'analyse des données et de production de diagnosituqe mis en communs (ESMValTool par exemple) pour garantir la reproductibilité des anlyses du GIEC.
- Évolutions/travaux en cours
- Des problèmes de reproductibilité computationnelle spécifiques: simulations checkpointées avec reproductibilité bit à bit car calcul des trajectoires conduites sur plusieurs mois. On vérifie les trajectoires au fur et à mesure pour repérer celles qui sont bizarres. Il se peut que l'env du calculateur évolue et on le détecte/on en tient compte. Vérification de la cohérence entre les différentes machines.
- Un jeu d'XP en 2010 (note technique de Servonnat et al. en 2013) avec le même code et les mêmes CI/CL tournées sur des calculateurs différents. Comme c'est chaotique, on a des trajectoires différentes immédiatement mais cohérence sur le plan du climat alors que quand on change les CI, les trajectoires sont très différentes.
### Raphaël Lévy (Université Sorbonne Paris Nord) Répliquer pour éclaircir une controverse scientifique : quelques enseignements d'une expérience en cours...
- Un sytème qui valorise certaines valeurs et certains comportements, mais au fond, des problématiques éthiques et sources d'injonctions contradictoires.
- Au sujet de points abordés hier et ce matin:
- Causes de la non reproductibilité: experimental design, manque de formation, Questionable Research Practives
- Communication avec le grand public sur les questions vives, et comment
- Ce qu'il y a dans les registered reports, c'est "digne de confiance" ? Non, c'est mieux mais il faut reconnaître qu'il y aura des problèmes. Si ça devient la mode et ce qu'il faut faire, on aura des problèmes. C'est **un moyen** intéressant.
- Les industries pharmaceutiques: good guys/bad guys ? Un peu simpliste, non ?
- Fabrique de l'ignorance. Récemment : [How an opioid giant deployed a playbook for moulding doctor's minds](https://www.bmj.com/content/385/bmj.q1208)
- Bulle scientifique $\sim$ bulle spéculative, d'espors, d'investissements.
- Ex: BioNano/COVID, IA, IRM pour Parkinson (des promesses, des centaines d'articles mais toujours pas d'applications cliniques),
- Conséquences des bulles: gâchis de ressources, amplification de recherche non-reproductible, erreur/désinformation scientifique/perte de confiance. La science s'auto-corrige-t-elle?
- Exemple des nanoparticules "zébrée" -> artefact de la méthode de mesure. [Stripy nanoparticles revisited](https://raphazlab.wordpress.com/2012/11/23/stripy-nanoparticles-revisited/)
- PubPeer: "online journal club" pour discussion d'article et fréquement autopsie des données et résultats
- NanoBubbles: projet ERC sur processus de correction scientifique en nanosciences. Voir https://nanobubbles.hypotheses.org/.
- Projet de réplication avec nanosondes
- Question: les nanoP peuvent-elles échapper aux endosomes? Pas par diffusion passive en tout cas car membrane = très imperméable -> endocytose = mécanisme le + probable
- Réplication de [Carbon-Dot-Based-Dual-Emission Nanohybrid Produces Ratiometric Fluorescent Sensor for In Vivo Imaging of Cellular Copper Ions](https://doi.org/10.1002/anie.201109089)
- Registered Report compliqué car article non classique, informations manquantes dans publis source et manque de réctivité de la part de l'équipe de recherche ayant publié l'article initial
- Abérrations dans les données sources détéctées (courbes avec "scale factor") -> continuation du projet avortée
- 2eme réplications sur [Nano-Flares: Probes for Transfection and mRNA Detetction in Living Cells](https://doi.org/10.1021/ja0776529)
- RR passe au stade 2 (In Principle Acceptance)
#### Questions
- Nous sommes effectivement tou·tes dans des bulles. Comment aller chercher des personnes qui ne sont pas déjà convaincues?
- Question politique sur comment on veut organiser la science. Transformation des pratiques par une société savante. On change en faisant du bruit. Un article avec 300 co-auteurs qui explique qu'il y a des problèmes d e reproductibilité, ça a du poids. Il faut aussi parler avec les personnnes influentes dans nos disciplines.
## 12h30-14h00 Pause déjeuner
## 14h00-17h00 Session 6 : Bilan et perspectives du réseau
### Organisation interne du Réseau
- Objectifs
- Identifiers les freins à la reproductibilité
- Faire évaluer les pratiques pour augmenter l'efficacité (la transparence et la confiance en) de la recherche.
****
##### GT - Logiciels :
Appel à contributions et/ou relecture, commentaires, suggestions ...
- https://gricad-gitlab.univ-grenoble-alpes.fr/gt-env-logiciels/sandbox-notecards/-/issues
- https://gt-env-logiciels.gricad-pages.univ-grenoble-alpes.fr/sandbox-notecards/
### Abel Brodeur (Université d'Ottawa, Institute for Replication, Canada) : Mass Reproducibility, the Institute for Replication and the Replication Games
### Gilles Mathieu (MESR Paris) (Isabelle Blanc excusée) : Bilan et clôture des journées
 Sign in with Wallet
Sign in with Wallet

