# OpenBioBlitz
Lien court: [frama.link/openbioblitz](https://frama.link/openbioblitz)
_Permettre par le numérique la contribution et la compréhension des citoyens pour le rassemblement des données de biodiversité lors d'un[ ](https://fr.wikipedia.org/wiki/BioBlitz)<u>BioBlitz</u>_
**Liens à regarder en priorité**
[](https://framindmap.org/c/maps/320935/)https://framindmap.org/c/maps/320935/
[](https://drive.google.com/drive/folders/0BxDsyq4MU1Ozem1XMEtHbnhhZnM?usp=sharing)[https://drive.google.com/drive/folders/0BxDsyq4MU1Ozem1XMEtHbnhhZnM?usp=sharing](https://drive.google.com/drive/folders/0BxDsyq4MU1Ozem1XMEtHbnhhZnM?usp=sharing)
Dépôt GitHub [](https://github.com/OpenBioBlitz)https://github.com/OpenBioBlitz
Futur site web sous github.io : [](https://openbioblitz.github.io/)[https://openbioblitz.github.io/](https://openbioblitz.github.io/OpenBioBlitz/)
**Définition BioBlitz**
Un BioBlitz est une étude sur une portion bien précise de terrain, où un groupe de scientifiques et/ou de bénévoles mènent un inventaire biologique intensif pendant un temps donné (en général de 24 à 48h). L'objectif est d'identifier et de répertorier toutes les espèces présentes dans une zone donnée.
**<u>PITCH:</u>**
Nous développons une application permettant d'inventorier et d'identifier les différentes espèces présentes dans un lieu donné, dans le cadre d'un rassemblement de naturalistes accessible à tous les publics qu'ils soient débutants ou experts. Accessible en version Online ou Offline.
**Ce qu'on propose**
Outils de récupération de données pour bioblitz et non de visualisation cartographique avec 3 niveau de récupération: débutant / intermédiaire / confirmé
Toutes les données sont automatiquement générées en standard international - [DarwinCore](http://www.canadensys.net/publication/darwin-core?lang=fr) - [](http://rs.tdwg.org/dwc/)http://rs.tdwg.org/dwc/
et complétées via des sites OpenSource comme Catalogue Of Life, Open Tree Of Life, Open Street Map
**Document de partage DRIVE**
Dans le Drive, se trouve:
* un jeu de données .txt : OntatioBioBlitz
* un jeu de données .txt BioBlitzUrbain
* Un article scientifique avec jeu de données tiré d'un BioBlitz
* les termes [DarwinCore](http://www.canadensys.net/publication/darwin-core?lang=fr) les plus importants en .doc
* un framework/template au niveau techno, app, termes, concept, etc. pour le projet
**Contribution en amont**
Jérémy Goimard
Carole Sinou
**Samedi **
Présents:
* Muriel Guernion
* Virginie / s'occupe de la documentation générale Hackpad + approche graphique
* Vincent Lasseur et Olivier Norvez / s'occupent de faire la "traduction" des termes de DWC en lexique commun accessible
* Julien / s'occupe de la programmation du formulaire de base
**Dimanche**
* [Olivier Norvez](/ep/profile/v3buVBuVVtf)
* Yohann Reverdy
* Vincent Lasseur
* virginie (à distance)
**<u>> contexte, "To do List"</u>**
* conceptualisation de l'interface et du déroulé des étapes de référencement
Idées et discussions en vrac de ce matin pour poser les objectifs à atteindre:
Idée de facilitation entre diff app et services rendus par INPN et GBIF (listing de données sur un espace/temps) et GBIF
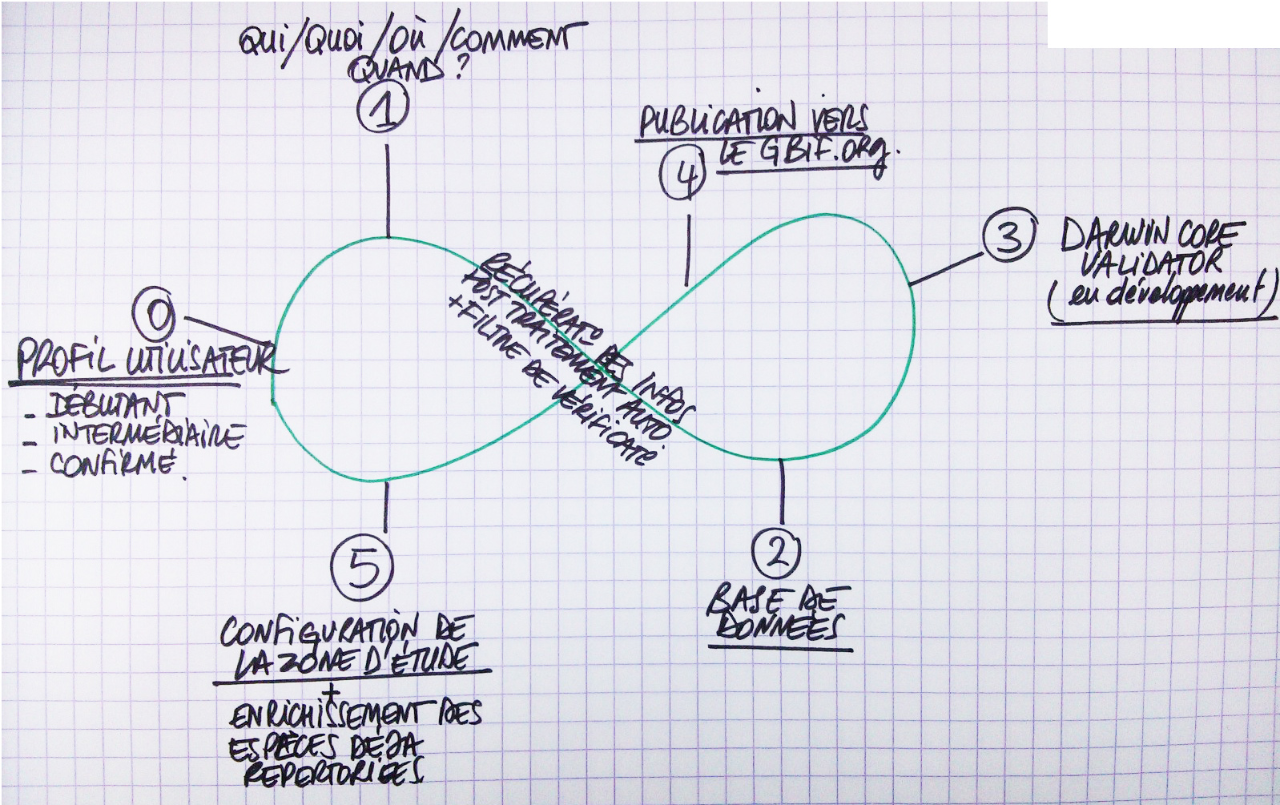
<u>Comment se fait un BioBlitz:</u>
1-définition de la zone géographique et durée de l'action
2-définition des acteurs et des parties-prenantes
3-identification de la typologie et méthodologie du Bio Blitz (Bio Blitz sur oiseaux / toute forme de vie / de zone géographique ...)
**<u>Liens Base de données:</u>**
-Taxonomie: Open tree of life / Catalogue of life
**<u>1/CONTENU DE L'INTERFACE UTILISATEUR:</u>**
**QUI: Identification PROFIL UTILISATEUR**
Débutant / Grand public : code couleur ? (vert clair)
Intermédiaire : code couleur ? (vert moyen)
Expert: code couleur ? (vert foncé)
**Quoi :ESPECE**
* nom commun , vernaculaire + proposition de noms spécifiques connus "locaux" déjà intégrés à la base /à enrichir au fur et a mesure des vérifications (par les scientifiques)
* nom scientifique
* taxonomie
* Occurrences
* Auto
* visuel de référence
**Où:** ** LOCALISATION GÉOGRAPHIQUE**
* Open street map pour localisation
**Quand : **
**DATE **:** **2017-03-04
**HEURE **Timer
**Comment:** **ACQUISITION DE LA DONNÉE**
* méthode d'acquisition de la donnée - typologie et mode d'observation - voir les valeurs possibles du champs DWC
observation à la jumelle ...
**DEFINITION DE LA TRAME DE BASE:**
_<u>1/Débutant: </u>_
Règne (animal, végétal, champignon)
Nom commun
Observé par
Description
Ville
Localisation
Remarques sur la localisation
Date
Précisions de date
Identifié par
Photo (à prendre si possible)
<u>Proposition de visuel possible pour le formulaire version Tablette:</u>

NB:
-il manque la case "**heure**" après la date pour indications complémentaires
-il manque la case "**Validation**" en bas de page (ou "suivant" pour passer à la page N°...)
**Sortie en public:**
- Pourquoi: valider ou non si nos champs descriptifs étaient pertinents ou non pour un enfant ou du-moins un public "débutant". cf. ci-dessus
* Méthodologie: on est sortis en ville (pratique car nous étions en centre-ville, pour aller demander à des personnes dans la rue
- Lieu: Espace des Sciences, Rennes
- Quand: Samedi à 17h45-18h
* Ce qu'il en est ressorti: Les termes et informations que nous devons identifier et comment les présenter doivent être sous forme de questions: ex. "Comment t'appelles-tu?" équivalent de recordedBy / Où te trouves-tu? = locality . etc. pour être bien comprises par les utilisateurs.
**Que vois-tu ?** (au choix : animal, végétal, champignion)
**Comment s'appelle cette espèce ?**
**Comment t'appelles-tu ?** (nom de la personne, nom de l'institution...)
**Pourrais-tu décrire cette espèce ?** (taille, couleur(s), comportement)
**Dans quelle ville as-tu vu cette espèce ?**
**Où as-tu vu cette espèce ?** (lieu-dit, arrondissement, nom de forêt ou rivière...)
**Que peux-tu me dire d'autre sur l'endroit ?** (montagne, fin de route, parking, terrain inondé...)
**Quelle est la date d'aujourd'hui ?** (année-mois-jour)
**Peux-tu préciser le moment de la journée ?** (lever du jour, crépuscule...) ou indication de l'heure?
**As-tu eu de l'aide pour identifier cette espèce ?**
**Si oui, comment s'appelle t'elle ? **(nom de la personne, nom de l'institution...)
+ **Photo** (à prendre si possible)
Print-screen de l'Arborescence pour débutant

Print-screen du de l'Arborescence DarwinCore

Print-screen de l'arborescence Exemple 1 utilisateur

Print-screen de l'arborescence Exemple 2 utilisateur

**Visio avec Marie-Elise Lecoq du GBIF-France**
- Olivier: y a t il un intérêt scientifique au projet?
- MEL : oui surtout pour le mode offline (ex. en Afrique où il y a une forte demande)
- Y t il des précédents dans les solutions déjà existantes (ex. i-Naturalist, i_record, etc.)?
- MEL: À voir avec i-Naturalist et WeDidBio si leur framework bien que répondant à des questions diff, apportent des solutions compatibles avec nos besoins.
- Sur le plan fonctionnel: faut-il laisser des champs libres ou restreindre les champs sous forme de listes?
- MEL: Les débutant pourraient préférer des listes à cocher et les confirmés des champs libres à remplir
D'un point de vue prog, il faut commencer par un nombre restreint de question, tant qu'on s'adresse à un public "non confirmé"
=> l'idéal serait qu'on pousse ce weekend la réflexion de l'interface des 3 niveaux utilisateurs jusqu'au bout afin de préparer le travail à faire (DB <->API <-> Interface) pour les prog-dev.
> **<u>Création compte GitHub</u>** > [](https://github.com/OpenBioBlitz)https://github.com/OpenBioBlitz
**Programmation**
1ere étape : installation & configuration IDE
* Pour développer une app Android, il nous un environnement de développement, avec tout les outils, c'est à dire un IDE. A priori **android studio** semble le plus simple à installer et utiliser.
* sudo add-apt-repository ppa:webupd8team/java
* sudo apt-get update
* sudo apt-get install oracle-java8-installer
2ème étape : creation d'un 1er projet
Impossible d'avoir une 1ere compile
* Gradle sync failed: Process 'command '/usr/local/jre/bin/java'' finished with non-zero exit value 2
cf. maxlath.eu/
**Dimanche matin**
[Yohann Reverdy](/ep/profile/njUv6VOEAeM) (développeur Web PHP spécialisé plus traitement de données bdd/API que interface) appropriation du projet.
constat : une partie Création de l'évènement (cf. Etape 0) et une partie "Répertorier"
besoin hors ligne : cf. pour des zones non "connectées"
besoin de valider Formulaire de base (champs + type de données), noms des champs par type de formulaire (débutant, etc.)
1er test/proto : création d'un formulaire test de saisie via framaforms [](https://framaforms.org/openbioblitz-1488706281)https://framaforms.org/openbioblitz-1488706281), les données saisies pourront ensuite être exportées et donc être exploitées pour envoi vers une base de données.
Constat : framaforms par facile à utiliser par rapport à un google forms...
Format d'export pas très user friendly (.tsv)
Code d'intégration :
<iframe src="[](http://framaforms.org/openbioblitz-1488706281)http://framaforms.org/openbioblitz-1488706281" width="100%" height="800" border="0" ></iframe>
A définir pour chaque formulaire (avec exemple) :
- description du formulaire
- libellé champ
- nom du champ (pour export texte)
- est-il obligatoire ?
- description
- type de données (texte, texte long, etc.)
- règle de validation
- préremplissage à partir d'une source extérieur ?
Remarques sur le formulaire :
- "localité" peut porter à confusion
- différence "remarques" vs "précisions"
- éviter au max. les champs libres
- prévoir des libellés de champs user friendly
### Vendredi 26 mai 2017 ###
Lieu MCE
support de documentation https://mensuel.framapad.org/p/miniNCC
### Samedi 9 septembre 2017
Idées développées par Gaetan D. :
Comme promis vous trouverez joint à ce message une proposition de Modèle Conceptuel de Données (a.k.a ERD Entity Relationship Diagram) pour le projet OpenBioblitz.
@jeremy, je te rejoins sur l'amélioration de la conceptualisation et je ne pense pas que ce soit trop scolaire de bien préparer ce sujet.
J'ajouterai même qu'une réflexion approfondie sur l'expérience utilisateur est également un préalable à la conception de ces formulaires. L'écriture de ce scenarii d'utilisation me semble un préalable à la conception de maquettes (si vous avez l'énergie de vous lancer dedans) et à une majorité des développements frontend (web et mobile).
Néanmoins, rien ne nous empêche d'avancer ensemble sur les développements de l'API et sur la conception d'une landing page du projet dès que l'ERD sera consolidé.
Quelques éléments techniques sur la structure du projet que j'ai envisagé:
Le language principal du backend seraitRuby.
La base de données principale du projet est PostgreSQL (version 9.6) pour laquelle j'aurais activé plusieurs extensions dont PostGIS (^_^), OpenFTS (pour la recherche provisoire, avant de passer à index Elasticsearch ou Algolia), HStore... Il est probable que j'utilise au maximum de leur possibilité la richesse des datatypes de PostgreSQL (JSON, Array...) ce qui potentiellement rend impossible toute migration vers un autre type de base de données relationnelles (MySQL, SQLite..).
Il y aura également des bases de données secondaires en particulier Redis qui servira de support pour l'éventuelle planification de traitement en arrière-plan (background jobs scheduling): post-traitement après chargement de médias, préparation des export DwCA, envoi en masse de courriers électroniques...
Le backend exposera 2 API, un peu à la Github (https://developer.github.com/):
une API de type REST, principalement pour les développeurs extérieurs
une API de type GraphQL, c'est l'avenir! perso j'aurais tendance à privilégier cette interface (je ne suis pas un expert GraphQL mais on apprend en faisant, non?)
Pour le front, le langage principal sera Javascript (je sais tout l'actuel engouement pour Typescript, mais personnellement je suis resté sur EcmaScript6/7) et le framework envisagé est React avec tout l'écosystème Redux, en particulier Redux Offline (https://github.com/jevakallio/redux-offline) pour la partie déconnecté(pour moi ce sera une première expérience).
@olivier, je ne sais pas si tu as envisagé cette option mais il est possible de structurer l'architecture du projet pour la rendre multitenant (désolé je ne sais pas comment traduire ça). C'est une architecture qui permet de répliquer le site sur différents schémas de bases de données et authentifier les utilisateurs sur différents sous-domaines tout en conservant l'ensemble des fonctionnalités et de la structure du site de base. Ex: lebiome.openbioblitz.cc, naturalistesenlutte.openbioblitz.cc... Celà permet aux utilisateurs de créer et gérer leur communauté de manière encapsulée. Tu retrouves ce type d'architecture sur des sites comme Basecamp ou Slack, pour ne citer qu'eux. Ça va à l'encontre d'une communauté globale OpenBioblitz mais ça peut avoir son intérêt... Vous me direz ce que vous en pensez.
Quelques informations complémentaires sur l'ERD.
Rien de folichon, j'ai repris les principaux champs des spécifications DwCA qui me semblait utile au projet. Mais je n'ai quasiment aucune expérience avec ces spécifications. Alors votre regard me sera très utile!
Les intitulés des champs utilise le format snake_case car les champs des tables PostgreSQL sont, il me semble, case insensitive. Bien entendu les API pourront exposer au format camelCase afin de respecter les standards DwCA.
J'ai pris la liberté de créer une table Organizations car je crois comprendre que les Institutions sont réservés aux dépositaires de collections. En utilisant les ressources documentaires du GBIF, j'ai pris connaissance du Global Registry of Biodiversity Repositories (http://grbio.org/). Savez-vous s'il existe un registre similaire pour les organisations qui supervisent des campagnes de relevés sur le terrain ou des observatoires de biodiversité? Cette table vise à compenser le non-succès dans mes recherches mais pourra être supprimée s'il existe un registre de référence reconnu par le GBIF dans ce domaine.
N.B: il existe une seule table Events mais l'application pourra gérer plusieurs type d'événements. Je comptais utiliser une technique appelée Single Table Inheritance (https://en.wikipedia.org/wiki/Single_Table_Inheritance), pour structurer différents Objets Ruby (enfin Rails pour être précis) dans l'application à partir d'une seule et même table. Voici les types d'objets que j'envisageais (intitulés non définitifs, et mobilisés par l'application / aucun lien avec les standards DwCA):
SurveyZones, sorte de hotspots de suivi. Peut accueillir plusieurs Bioblitzes (voir ci-après). Pas d'événement parent. Associées à un groupe de participants/volontaires. Gérées par un ou plusieurs coordinateurs. Objectif: Faire un focus sur une zone à cartographier en priorité (source d'inspiration: Disaster Mapping Projects de Humanitarian Openstreetmap Team). Facultatif. Pas d'occurence associée.
Bioblitzes, regroupement éphémère de participants/volontaires pour inventorier et cartographier la biodiversité d'un site. Peut-être un enfant d'une SurveyZone ou être orphelin. Associées à un groupe de participants/volontaires. Gérées par un ou plusieurs coordinateurs. Recommandé. Occurences associées possibles
SamplingEvents, session d'observation de biodiversité d'un participant/volontaire. Peut-être un enfant d'une SurveyZone, d'un Bioblitz ou être orphelin. Associé à un participant (ne figure pas sur l'ERD). Requis. Occurrences associées recommandées
[ Est-ce que cette conceptualisation fait sens pour l'application? Ou suis-je à côté de la plaque? ]
La gestion des utilisateurs (je vous passe mes tergiversations sur l'authentification: JWT, OAuth, Passwordless authentification...). Voici la proposition: En gros, un utilisateur peut définir un type de profil (débutant, amateur, expert) qui conditionnera le type de formulaire qui lui seront proposé par défaut dans l'application. Un utilisateur peut être membre/référents (Member) d'une ou plusieurs organisations (Organizations) et ainsi bénéficier des notifications (email, push...) transmises par les référents de cette organisation. Un utilisateur peut être participant/coordinateur (Attendee) d'un ou plusieurs événement (Events) hors SamplingEvents qui sont toujours associés à un utilisateur.
Les Metadata. Si j'ai bien compris ces metadonnées sont associées à la publication d'un jeu de données candidat à une publication, n'est-ce pas? Dans ce cas, les référents(SurveyZone) /coordinateurs(Bioblitz) / propriétaires(SamplingEvent) peuvent définir les metadata de leurs événements. Les occurrences ne seront publiés que par l'intermédiaire de la publication d'un événement: par dépendance, les occurrences auront donc toujours des metadonnées associées (le champ eventID de la table Occurences sera requis avant validation de l'enregistrement).
Pas de table Taxon dans cet ERD: l'idée est de soumettre des requêtes à des services de données extérieurs (EOL, GBIF Backbone Taxonomy...) et de récupérer uniquement le nom scientifique et l'identifiant du-dit taxon dans la table Occurrence. Celà dit, si on veut fonctionner en mode déconnecté, il faudra bien mettre en cache une base de référence mais c'est un autre sujet.
J'ai ajouté mais pas détaillé des tables pour la gestion des médias associés, la gestion de l'identification collaborative. Personnellement (même si je trouve ça très intéressant et même essentiel), je ne le mettrais pas à l'agenda des premiers développements.
--------------------------------------------------------------
**LICENCE**

OpenBioBlitz de [OpenBiolBlitz et contributeurs hackpad](https://github.com/OpenBioBlitz) est mis à disposition selon les termes de la [licence Creative Commons Attribution 4.0 International](http://creativecommons.org/licenses/by/4.0/).
Fondé(e) sur une œuvre à [OpenBioBlitz](/jVIIVR11rac).
 Sign in with Wallet
Sign in with Wallet

