全基因體定序(WGS)
===
###### tags: `基因體`
###### tags: `基因體`, `WGS`
<br>
[TOC]
<br>
## WGS
- ### 全名:
- Whole Genome Sequencing,全基因體定序
- ### 定義:
- 對「單一 生物體 基因組」解析「完整 DNA 序列」的過程
- 定序:將 DNA 化學分子,轉錄成 A/T/C/G 文字檔

([圖片來源](https://zh.wikipedia.org/wiki/%E4%BA%BA%E9%A1%9E%E5%9F%BA%E5%9B%A0%E7%B5%84))
- WGS 包含
- 生物體的染色體 DNA
- 粒線體 DNA
- 植物葉綠體 DNA
- 應該是指
- 動物:動物染色體 DNA + 粒線體 DNA
- 植物:植物染色體 DNA + 粒線體 DNA + 葉綠體 DNA
<br>

([圖片來源](https://kongtai.com.tw/%E7%B4%B0%E8%83%9E%E7%99%82%E6%B3%95/%E7%B2%92%E7%B7%9A%E9%AB%94))
- ### 定序目的:
- 研究「基因」與「疾病」的關係
- 因為 →「疾病」可能發生在有問題的基因序列
- 所以 → 找出基因序列變異的地方,又稱:
- Variant detection (變異點偵測)
- Variant calling (變異點偵測)
- #### 遺傳變異種類:

([圖片來源](http://www.yourgene.com.tw/content/messagess/contents/655406000314624540/))
- [單一核苷酸多型性 (Single Nucleotide Polymorphisms, SNPs)](https://unclegene6666.pixnet.net/blog/post/308333779)
- 發音:[snips](http://www.tma.tw/ltk/100540603.pdf)
- 定義:大多數是 1bp 的變異
- [插入與缺失 (Insertions and Deletions, INDELs)](https://www.scmh.org.tw/internet/blog/Blog_Preview.aspx?Blog=Q0FfREVQVDAwMQ==&Article=48807515-ca94-47b6-8f85-5832b9b78d5e)
- 定義:小於 50 bp 的變異
- 染色體結構變異 (Structure Variations, SVs)
- 定義:50 bp 以上的變異
- 長片段序列插入或刪除(Big Indel)
- 串連重複(Tandem Repeate)
- 反轉(Inversion)
- 易位(Translocation)
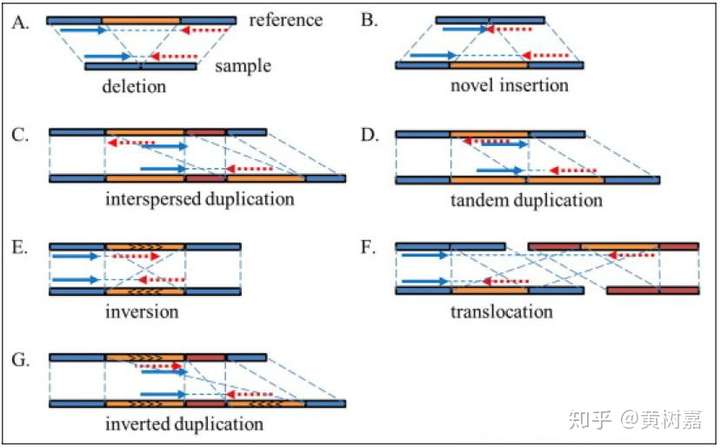
([圖片來源](https://zhuanlan.zhihu.com/p/40290546))
<br />

「9號染色體」與「22號染色體」發生易位,形成費城染色體
([圖片來源](https://zhuanlan.zhihu.com/p/40290546))
- [相關檢測工具超過 130 種](http://www.huangshujia.me/2018/07/22/2018-07-22-Introduction-the-detection-of-structure-variants.html)
- 拷貝數變異 (Copy Number Variations, CNVs)

([圖片來源](https://en.wikipedia.org/wiki/Copy-number_variation))
- 用語:
- 台灣:複製數變異
- 大陸:拷貝數變異
- 定義:1kb ~ 3MB(3000kb) 的變異
- [屬於 INDELs,但往往序列很長,特別獨立分類](http://www.huangshujia.me/2018/07/22/2018-07-22-Introduction-the-detection-of-structure-variants.html)
- ...等等 (DNA甲基化的表觀遺傳學變異)
- ### 過去計畫&計畫中
- #### 人類基因體計畫(The Human Genome Project, HGP) ([wiki](https://zh.wikipedia.org/wiki/%E4%BA%BA%E7%B1%BB%E5%9F%BA%E5%9B%A0%E7%BB%84%E8%AE%A1%E5%88%92))
- **目標**
- 對人類全基因體進行解碼
- **時間**
- [1990 年開始,於 2003 年完成](http://www.art.ntou.edu.tw/masters/download/10602/0607PDF.pdf)
- 完成度92%,詳見 [wiki](https://zh.wikipedia.org/wiki/%E4%BA%BA%E7%B1%BB%E5%9F%BA%E5%9B%A0%E7%BB%84%E8%AE%A1%E5%88%92)
- 基因組捐獻者
- 兩名男性和兩名女性
- 從他們血液中的白血球,分離出的 DNA
- **花費**
- [85億美金](https://news.gbimonthly.com/tw/magazine/article_show.php?num=42339&page=2044&range=news)
- **成果**
- 版本演進說明:
- [GRCh37 (2009.02.27)](https://www.ncbi.nlm.nih.gov/assembly/GCF_000001405.13/),又稱 hg19
- GRCh37 全名
- [Genome Reference Consortium Human Build 37](https://www.ncbi.nlm.nih.gov/assembly/GCF_000001405.13/)
- 基因體參考序列聯盟-人類基因體37
- [b37](https://blog.csdn.net/u014182497/article/details/84032261) 是 GRCh37 的升級板
- 命名規範、座標系統規範
- 粒線體和開頭沒定序到的基因序列
- [hs37d5 (b37+decoy)](https://blog.csdn.net/u014182497/article/details/84032261) 是 b37 的升級板
- 增加一條病毒序列(皰疹病毒)
- 補充:[英文定義](http://www.imsbio.co.jp/RGM/R_rdfile?f=BSgenome.Hsapiens.1000genomes.hs37d5/man/package.Rd&d=R_BC)
> Full 1000genomes Phase2 Reference Genome Sequence (hs37d5), based on NCBI GRCh37.
- [GRCh38 (2013.12.17)](https://www.ncbi.nlm.nih.gov/assembly/GCF_000001405.26/),又稱 hg38
- [GRCh38.p12 (2017.12.21)](https://www.ncbi.nlm.nih.gov/assembly/GCF_000001405.38) (patch)
- [GRCh38.p13 (2019.02.18)](https://www.ncbi.nlm.nih.gov/assembly/GCF_000001405.39) (patch)
- 補充:[UCSC 提供的資訊](https://genome.ucsc.edu/cgi-bin/hgTables?clade=mammal&org=Human&db=hg19)
- Dec. 2013 (GRCh38/hg38)
- Feb. 2009 (GRCh37/hg19)
- Mar. 2006 (NCBI36/hg18)
- May 2004 (NCBI35/hg17)
- July 2003 (NCBI34/hg16)
- [參考基因體-版本對照](http://www.bio-info-trainee.com/1469.html)
| NCBI | UCSC | Ensembl | 年代 |
| ------ | ---- | -------- | ---- |
| GRCh38 | hg38 | release_76/77/78/80/81/82 | 2013/12 |
| GRCh37 | hg19 | release_59/61/64/68/69/75 | 2009/02 |
| GRCh36 | hg18 | release_52 | 2006/03 |
- [多種版本同時並行,我究竟該用哪個版本呢?](http://toolsbiotech.blog.fc2.com/blog-entry-119.html?sp)
- 參考基因體
- 利用來自”多個”DNA提供者的基因體,進行定序之後而組裝而成的,因此不能準確地代表任何一個人的基因體序列。
- [Broad Institude 針對各版本的說明](https://gatk.broadinstitute.org/hc/en-us/articles/360035890711-GRCh37-hg19-b37-humanG1Kv37-Human-Reference-Discrepancies#b37)
- [hg19](https://gatk.broadinstitute.org/hc/en-us/articles/360035890711-GRCh37-hg19-b37-humanG1Kv37-Human-Reference-Discrepancies#hg19)
- [GRCh37](https://gatk.broadinstitute.org/hc/en-us/articles/360035890711-GRCh37-hg19-b37-humanG1Kv37-Human-Reference-Discrepancies#grch37)
- [b37](https://gatk.broadinstitute.org/hc/en-us/articles/360035890711-GRCh37-hg19-b37-humanG1Kv37-Human-Reference-Discrepancies#b37)
- [HumanG1Kv37](https://gatk.broadinstitute.org/hc/en-us/articles/360035890711-GRCh37-hg19-b37-humanG1Kv37-Human-Reference-Discrepancies#humanG1Kv37)
- ==[2020-01-15 了解人类不同版本参考基因组及如何选择](https://www.codenong.com/jse65115b4633a/)==
- **选择参考基因组的建议**
- 比对至GRCh37(hg19),使用hs37-1kg
- 比对至GRCh37,并且认为 decoy sequence* 有助于variant calling,使用hs37d5
- 关于decoy sequence,在博文《关于人参考基因组fasta文件的组成部分说明》中有提及,EB病毒基因组:
- https://www.jianshu.com/p/5b73773e30ef
- fasta組成
- Primary assembly
- Assembled chromosomes
- Unlocalized sequence
- Unplaced sequence
- Alternate contigs, alternate scaffolds或 alternate loci
- PAR 区域: 伪染色体序列(pseudoautosomal region)
- decoy基因组
- (誘餌基因組)
- BWA的發明家Heng Li創建了一個完整的“誘餌基因組”,以捕獲來自人類外顯子組或整個基因組測序中未能與人類參考基因組對齊的讀數。
- EBV基因組只有170kb,而誘餌的完整基因組只有36Mb。
- **[最新人類參考序列的兩個版本NCBI(GRCh38)和UCSC(hg38)有什麼差異?](https://kknews.cc/news/lngvmeg.html)**
1. UCSC的hg38相比於NCBI的GRCh38缺少chrEBV(Epstein-Barr virus)序列。EBV本身不是人所有的,但由於很多細胞系在培養的過程中都需要藉助EBV,因此對於許多通過細胞系測序而來的數據中(比如海拉細胞系),就會混有這個序列。在NCBI的新版本中已經加入EBV序列,但是UCSC並沒有相應的更新;
2. UCSC版本缺少decoy序列,這是參考序列中沒有,但卻是其他人群特有的人類序列,作為參考序列不應該缺少;另外還缺少HLA分型序列;
3. _
4. Y染色體上存在較大差別。NCBI上的GRCh38,在Y染色體上的兩個PAR區域(pseudoautosomal region,偽染色體區域)用N來代替了,而UCSC並沒有做類似的處理。這樣會導致它們在Y染色體序列中存在明顯的差異,進行序列比對時結果也會有所不同。
- **[在Long Ranger WGS中使用hs38d1誘餌重要嗎?](https://kb.10xgenomics.com/hc/en-us/articles/360001610751-Is-it-important-to-use-the-hs38d1-decoy-in-Long-Ranger-WGS-)**
- 在沒有誘餌的情況下,在裝配中對準的任何位置都將成為錯誤的位置,並可能導致假陽性。
- 包括誘餌序列也可以減少比對時間。來自誘餌代表的基因組區域的讀段將快速映射到誘餌,從而避免了浪費的計算能力
- 其實同樣版本 hg19,仍有可能有小版本差異
如 knownGene 和 refGene
- knownGene
[](https://i.imgur.com/ZgkQRa3.png)
- refGene
[](https://i.imgur.com/djX2rVI.png)
- diff

<br>
- DNA 資訊
- DNA 的 1.5%
- 蛋白質編碼基因 → 外顯子(exsome)
- DNA 的 98.5%
- 通知基因何時啟動和關閉的調節序列
- 會轉錄出 RNA ,但不會轉譯為蛋白質的基因
- DNA 序列長度
- **3,099,706,404**
≒ **3.09x10E9**
≒ **3.1x10E9**
- 有時會看到文章寫:
人類有30 (or 31)億個基因/鹼基對,都對
<br>
- 個體(人與人)之間的差異:
- [0.3% (約 900 萬鹼基對)](http://www.tma.tw/ltk/100540603.pdf)
- 0.1% ([圖片來源](https://twitter.com/genomicsedu/status/1165942537032519681))

- [群體(人與基因庫)之間的差異](http://www.tma.tw/ltk/100540603.pdf):99.7%
- [台灣人體資料庫](https://www.twbiobank.org.tw/new_web/news.php)

- [人類有三萬個基因](http://www.tma.tw/ltk/100540603.pdf)
>某些時候,基因會先支配合成 pre-mRNA 之分子,此 pre-mRNA 核苷酸序列中一片段含有至少一個intron或一個exon,其在接合(alternative splicing)過程會形成不同mRNA (differential RNA processing) ,因此合成不同的蛋白質。這正能解釋何以人類細胞約含有三萬個基因,但卻能製造二十萬~一百萬個不同的蛋白質之理由。
- [人類染色體版本釋疑](https://yourgene.pixnet.net/blog/post/117778207)
- [次世代定序知識櫥窗-人類參考基因體 (Human Reference Genome)](http://toolsbiotech.blog.fc2.com/blog-entry-119.html)
- 人類參考基因體對於研究人員來說,使用上有哪些要注意的呢?
- 多種版本同時並行,我究竟該用哪個版本呢?
- 那我該選擇哪個版本呢?是不是最新的版本最好?
- [G1k = 1000 genomes](https://www.biostars.org/p/13879/)
- 同一時間發表、不同名稱的人類參考基因體,那差別是在哪兒呢?
GRCh37 / hg19 / b37 / humanG1Kv37
- #### 千人基因體計畫(1000 Genomes Project)
- 計劃者
- International Genome Sample Resource (IGSR)
國際基因體樣本資源
- 最新版本
- GRCh38
- [1000 Genomes Browser](https://www.ncbi.nlm.nih.gov/variation/tools/1000genomes/)
- 資料來源
- ftp://ftp.1000genomes.ebi.ac.uk/vol1/ftp/pilot_data/data
- 2017, NCBI 新聞稿
- [The international Genome sample resource (IGSR): A worldwide collection of genome variation incorporating the 1000 Genomes Project data](https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5210610/)

- #### 跨組學精準醫學研究(Trans-Omics for Precision Medicine, TOPMed)
- 計劃者
- NIH(美國健康研究院)和其他
- 起始於
- 2015
- 參加者/志願者
- 目標:要收集到一百萬個志願者(?)
- 跨越很多年齡組,來自不同社會,文化和種族/民族背景
- 介紹
- [MESA 和精準醫學](https://www.mesa-nhlbi.org/publicdocs/Newsletter/21Ch_Winter2017.pdf)
- #### 臺灣人體生物資料庫
- [2017《Taiwan Biobank 臺灣人體資料庫》 動畫簡介 (3min)](https://youtu.be/a4yK1AhUrRw)
- 目標:現代醫學 → 個人醫學,達到疾病預防與精準醫療 → 更好的健康生活
- [參加流程](https://www.twbiobank.org.tw/new_web/join-flow-before.php)
- 相關文章
- [[從 DNA 看世界系列] 醫療除了「精準」,還需要「公平」](https://medium.com/@chungtsai/a0922675d272)
- ### 應用範圍:
- 預防醫學、疾病檢測 (事前)
- 產前檢查:
- [精神分裂症案例](https://youtu.be/x5aynJYhAXs?t=797)
- [羊膜穿刺需要做嗎?醫師教你自己判斷!](https://www.mombaby.com.tw/pregnacy/notes/articles/12782)
- 個人化醫療 (Personalized Medicine) (事後)
- 針對 DNA 下藥,減少不必要的醫療資源浪費
- 影片:
- [中天調查報告》預存健康不是夢?
基因檢測「算病」 預知疾病紀事 201504194](https://www.youtube.com/watch?v=Aj-wzRP5KxE)
- 人類癌症研究 (Cancer research)
- 全基因組關聯分析 (genome-wide association study, GWAS)
<br />
## [序列組裝](https://zh.wikipedia.org/wiki/%E5%BA%8F%E5%88%97%E7%B5%84%E8%A3%9D) (基因的拼接)
- ### De-novo 組裝
- De-novo:為拉丁語,意指「新的」
- 在沒有參考序列的情況下,僅使用序列片段來組裝的方法。
- Overlap Layout Consensus(OLC)法(重疊/配置/共識)

- De-Bruijn(DBG)法

<br />

([圖片來源](https://investigator.tw/7061/%E4%B8%89%E4%BB%A3%E5%AE%9A%E5%BA%8F%E5%86%8D%E5%BA%A6%E6%8E%80%E8%B5%B7%E9%9D%A9%E5%91%BD%EF%BC%9A%E5%BE%9E%E9%A0%AD%E5%AE%9A%E5%BA%8F%EF%BC%88de-novo-sequencing%EF%BC%89/)) 長序列定序有利於解決上述問題
<br />
- 重疊問題:

- 疾病可能剛好出現在不完整的序列上 (GRCh38 的缺陷)
- [顯性腎臟疾病 MCKD1](https://investigator.tw/1185/)
- ### Mapping 組裝
- 在有參考序列的情況下,將測序片段比對至參考序列上,以取得組裝結果。
- 類似概念:在拼拼圖的時候,需要將每一片拼圖與完成圖進行比對,才容易完成拼圖。
<br />
## 定序儀器 NGS (第二代定序儀器)
- ### 定序策略(定序演算法):
- 短序列 (Short-Reads)
- 雙末端(Paired-Ends)
- 示意圖一

([圖片來源](http://blog.sina.com.cn/s/blog_a893ace40101hum2.html))
- 示意圖二


([圖片來源](https://yourgene.pixnet.net/blog/post/68952443-paired-end-v.s.-single-end))
[透過 paired-ends 得到最好的序列組裝結果](https://yourgene.pixnet.net/blog/post/68952443-paired-end-v.s.-single-end)
- 不同長度的插入片段 (Insert-Size)
(將 3 種相互結合搭配來定序)
- ### 序列片段分佈:[負二項分布](https://kknews.cc/zh-tw/news/66y86eq.html)

([圖片來源](http://www.huangshujia.me/2018/07/22/2018-07-22-Introduction-the-detection-of-structure-variants.html))
- ### [Illumina 技術示意圖](https://hilelojack.pixnet.net/blog/post/99442019-%E4%BA%8C%E4%BB%A3%E6%AF%94%E4%B8%80%E4%BB%A3%E5%A5%BD%3F)

- 合成式定序(sequencing by synthesis)
- [[橋接的動畫] Intro to Sequencing by Synthesis: Industry-leading Data Quality](https://www.youtube.com/watch?v=HMyCqWhwB8E)
- [Illumina Sequencing by Synthesis](https://www.youtube.com/watch?v=fCd6B5HRaZ8)
- [Illumina Sequencing Technology](https://www.youtube.com/watch?v=womKfikWlxM)
- [Patterned Flow Cell Technology | Illumina Video](https://www.youtube.com/watch?v=pfZp5Vgsbw0)
- [【陈巍学基因】视频1:Illumina测序化学原理](https://www.youtube.com/watch?v=VvS8NEJGxnM)
- ### 品質問題 (Quality control, Quality checking, FastQC)
[參考此篇](/9iqEiCiNQbmbnRKbgqLcaA)
- ### NGS 的費用
- [如何詮釋基因上的變異─導讀ACMG與AMP](https://yourgene.pixnet.net/blog/post/115335385)
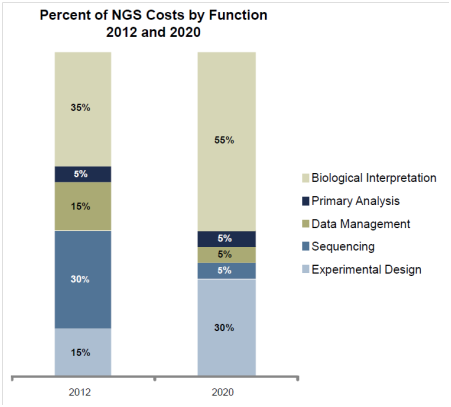
- ### 其他相關資料
- [簡介次世代定序技術及美國的法規管理 - 財團法人醫藥品查驗中心](https://www.cde.org.tw/Content/Files/Knowledge/45189bb7-1908-4797-a371-b0dee4d9fb53.pdf)
- 第一代定序
- 次世代定序
- 樣本庫製備
> 採集待測樣本並進行核酸萃取後,再透過基因工程的方法,將待定序的基因. 序列隨機切割成大小不等的短片段,此步驟稱為核酸片段化(fragmentation)。接著. 再將這些短片段基因序列的末端接上轉接子(adapter),以製成樣本庫。
- 兩代定序技術的差異
- [Illumina各型螢光頻率定序方式介紹](https://yourgene.pixnet.net/blog/post/118971582)
<br />
## 定序目標
- **全基因** -> 全基因組定序(Whole genome sequencing, WGS)
- **外顯子** -> 全外顯子定序(Whole Exome Sequencing, WES)
- **簡單講:「外顯子」就是指「能轉譯成蛋白質」的基因**
- [定序分析服務簡介](http://www.sequencing-tech.com/content/%E5%A4%96%E9%A1%AF%E5%AD%90%E5%BA%8F%E5%88%97%E5%88%86%E6%9E%90-exome-sequencing)
- 外顯子 = 蛋白質編碼區域 (protein-coding regions) 與非轉譯區域 (Untranslated Regions)
- 占人類基因體的 1%
- 能檢測到 99% 的 SNPs
- [外顯子組定序](http://scimonth.blogspot.com/2015/05/blog-post_22.html)
- 85%的遺傳疾病,都是由蛋白質的失常引起的
- 目的:
- 發掘新的致病基因
- 找出「患者有、而其他人沒有」的基因序列
- **特定目標基因** -> 目標區間定序(Targeted Sequencing)
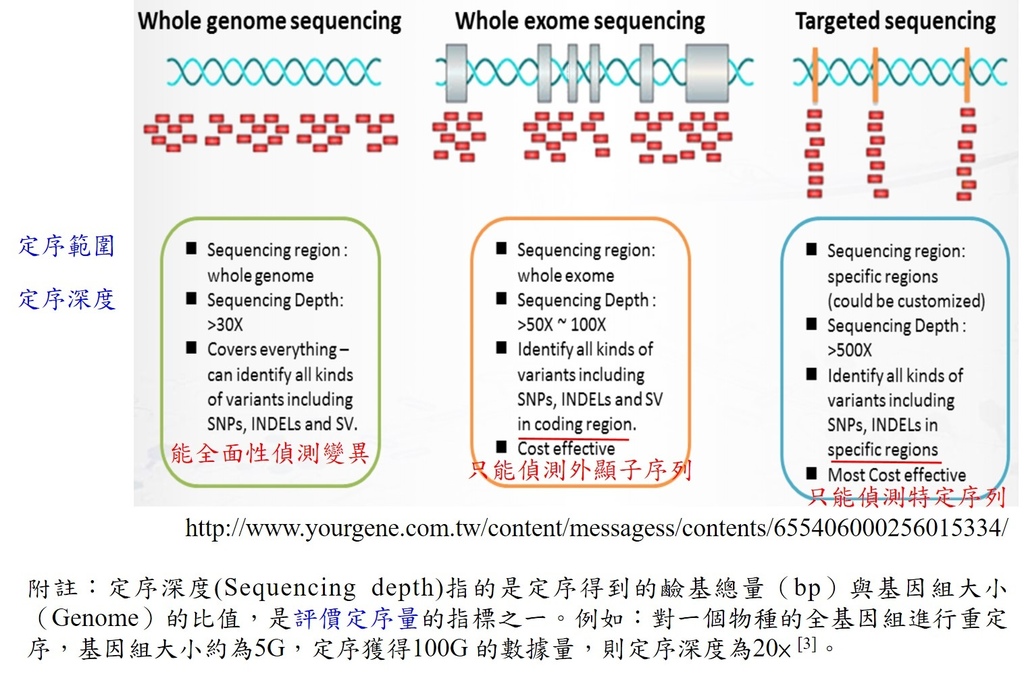
<br />
## 定序流程
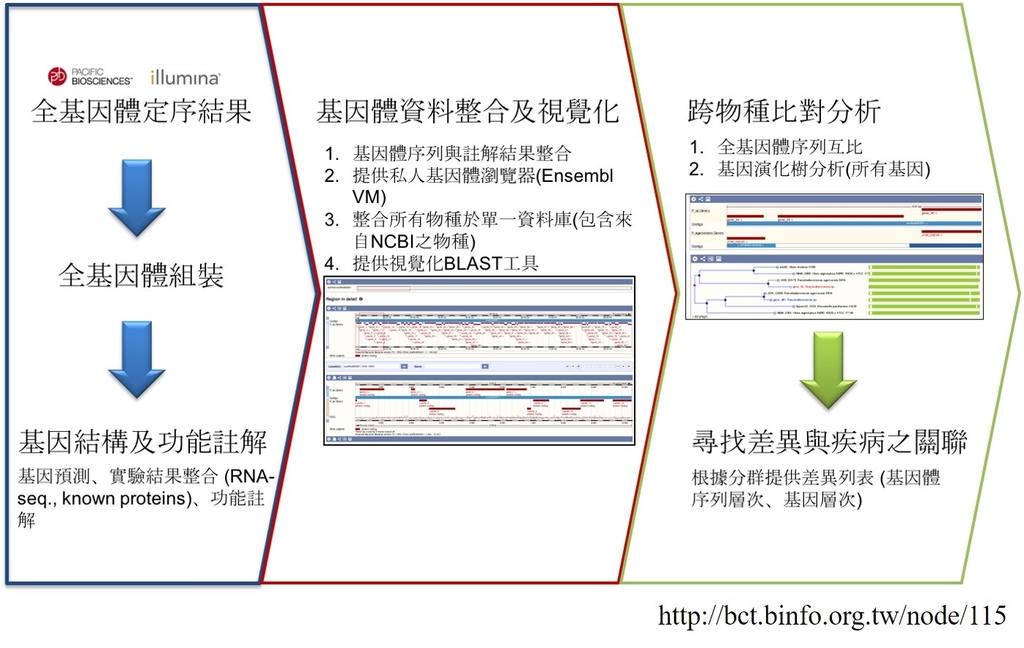
([圖片來源](https://unclegene6666.pixnet.net/blog/post/352053530))
<br/>
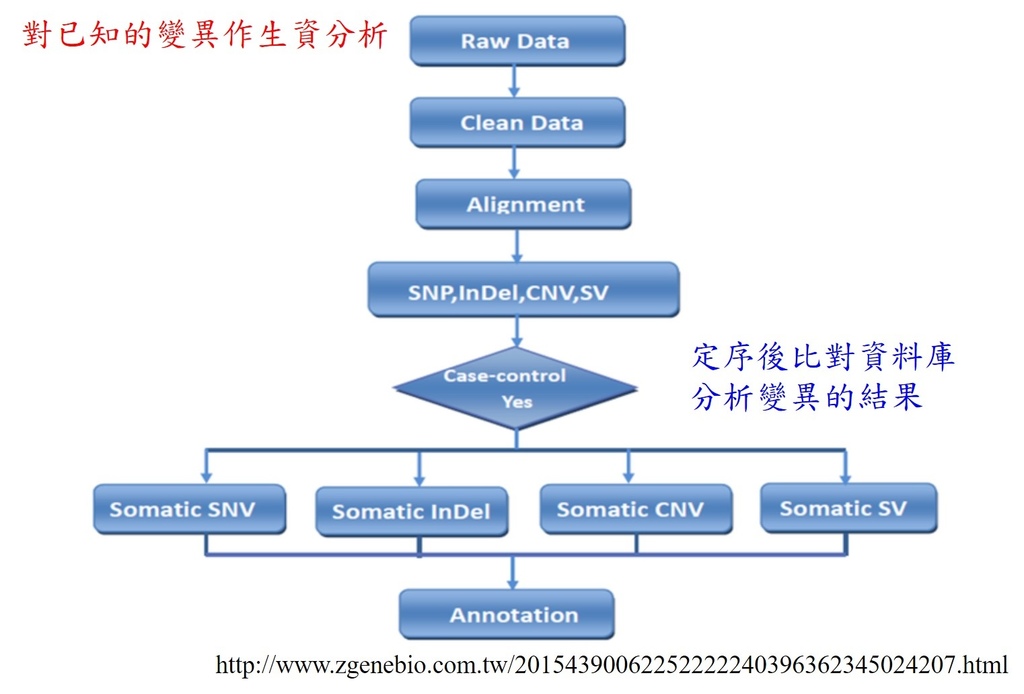
([圖片來源](https://unclegene6666.pixnet.net/blog/post/352053530))
<br />
## 尋找變異(variant discovery)
- 尋找變異是很困難的
- 如同大海撈針,無法確定「變異的基因」是否跟「疾病症狀」有相關
- 透過 Case-Control Study (病例對照研究法) 來尋找變異
- 通常藉由 **「病例組 (cases)」和「對照組 (controls)」** 交叉比對,尋找差異的基因
- [案例1](https://home.gamer.com.tw/creationDetail.php?sn=861126):HAR1基因 ([wiki](https://en.wikipedia.org/wiki/Human_accelerated_region_1))
> 人類和黑猩猩的最長相異序列(HAR1),長118個核酸,有18個鹼基不同。透過分子螢光標記,發現會活化某一類的神經元。這種神經元會影響大腦皮層皺摺;若神經元出錯,將導致平腦症。
- [案例2](https://home.gamer.com.tw/creationDetail.php?sn=861126):FOXP2基因
> 咬字說話的能力,它和語言有關。英國牛津大學科學家在2001年的報告指出,FOXP2 突變的人,儘管有處理語言的認知能力,但是臉部無法做出正常說話時所需的一些細微快速的動作。典型的人類 FOXP2 基因和黑猩猩的基因序列有幾個地方不同,其中兩個鹼基置換會改變蛋白質的胺基酸,其他置換則可能影響這個蛋白質作用的方式、時間與部位。
- [案例3](https://home.gamer.com.tw/creationDetail.php?sn=861126):ASPM基因 ([wiki](https://en.wikipedia.org/wiki/ASPM_(gene)))
> 在人類和其他動物腦容量的研究中,科學家透過分析腦容量縮小了70%的小腦症患者,發現了 ASPM 和其他三個基因 MCPH1、CDK5RAP2 和 CENPJ 控制了腦的大小。
- [案例4](https://youtu.be/x5aynJYhAXs?t=562)
> 同性戀基因 Xq28 是否存在?結果是誤判基因
- [科學人2009年第88期6月號:人為何為人](https://home.gamer.com.tw/creationDetail.php?sn=861126)
> 人類基因體中佔 98.5% 的非蛋白質編碼序列,這些「垃圾」序列如何變得越來越有價值?
<br />
## 術語
- 轉移至 [生物資訊.md](https://github.com/tsungjung411/language-study/blob/master/en/terms/生物資訊.md)
<br>
## 定序廠商
- [有勁基因體核心實驗室 - 定序委託單附件](file:///home/tj/Downloads/YG2-QC02-01_Request_attach-V2_2015.03_.26_.pdf)
<br>
## 參考資料
- 基因叔叔 / 基因世界
- [什麼是全基因定序(Whole genome sequence ,WGS)](https://unclegene6666.pixnet.net/blog/post/352053530)
- [什麼是單核苷酸多型性 (Single Nucleotide Polymorphism,簡稱SNP](https://unclegene6666.pixnet.net/blog/post/308333779)
- [一個基因突變的節奏—DNA損傷](https://unclegene6666.pixnet.net/blog/post/304711852)
- [基因體定序之現況與展望](http://bc.imb.sinica.edu.tw/images/biotech/14_16.pdf)
- 有勁基因
- [螢光定量PCR原理簡介](https://yourgene.pixnet.net/blog/post/119714034)
- [Optical mapping 技術簡介](https://yourgene.pixnet.net/blog/post/63708485)
- [De novo assembly──人類基因體補完計畫](https://investigator.tw/1185/)
- [科學人2009年第88期6月號:人為何為人](https://home.gamer.com.tw/creationDetail.php?sn=861126)
- [二代測序中Duplication rate 雜談(二)](https://read01.com/E8EyRa6.html)
- [一篇文章说清楚基因组结构性变异检测的方法](http://www.huangshujia.me/2018/07/22/2018-07-22-Introduction-the-detection-of-structure-variants.html)
- [从零开始完整学习全基因组测序数据分析:第4节 构建WGS主流程](https://mp.weixin.qq.com/s/awdjoXRYobrQAbXmAp3C0g)
<br />
<!--
## 心得感想
- 癌症
- **癌症起源**:
- DNA 損傷,來不及修復
- **萬惡之源**:
- 就是某個部位,持續發炎,就可能造成 基因損傷
- **基因與電腦的關係**:
- 基因損傷,就跟電腦程式有 bug 一樣(程式寫不好)
- 輕則當機,造成某種疾病
- 重則造成癌症,就跟電腦程式無止盡執行一樣 (迴圈沒有條件中止)
- **編碼方式**:
- 電腦程式是二進位編碼,由 0 和 1 組成
- 基因是四進位編碼,由 A / T / C / G 四種鹼基 組成
-->
 Sign in with Wallet
Sign in with Wallet
 Sign in with Wallet
Sign in with Wallet