假設腫瘤(tumors)的資料如下:
Total: 100
Total positive: 9 (9%)
Total negative: 91 (91%)
在不平衡數據集中,僅僅依賴準確率(accuracy)來評估模型性能是不夠精確的
偏向多數類別:
在不平衡數據集中,多數類別樣本數遠多於少數類別。由於模型在訓練過程中接觸到的多數類別樣本更多,它更有可能傾向於預測多數類別。這樣,即使模型大部分時間都預測為多數類別,也會有很高的準確率。
例如,在例子中,假設模型總是預測“沒有腫瘤”,那麼模型的準確率會是91%,但這個模型對於檢測腫瘤的病人(正類別)的能力非常差。
忽略少數類別的性能:
高準確率可能掩蓋了模型對少數類別(如腫瘤病人)的差勁表現。由於少數類別樣本數少,模型即使預測錯誤,也不會顯著影響整體準確率,但這種錯誤對實際應用影響很大,特別是在醫療診斷中。
模型效果的錯誤認知:
僅看準確率,容易對模型的實際效果產生錯誤認知,導致模型在應用中表現不佳,特別是當我們更關心少數類別的預測時。
EX:
實際類別 預測類別 數量
Positive Positive 1
Positive Negative 8
Negative Positive 0
Negative Negative 91
準確率:模型預測的準確率是92%,這看起來非常高,給人一種模型表現良好的錯覺。
精確率:模型對於預測為正類(腫瘤)的樣本中,實際為正類的比例是100%,因為模型只預測了1個腫瘤,且這個預測是正確的。
召回率:模型對於實際為正類(腫瘤)的樣本中,僅正確預測了11.1%,這表示模型對於腫瘤的檢測能力非常差。
實際應用影響
在醫療診斷中,如果模型不能有效地檢測出腫瘤患者(即召回率低),那麼即使模型的整體準確率很高,這個模型在實際應用中仍然是不可靠的。漏診的腫瘤患者(8個漏診的樣本)可能會錯過及時治療的機會,對病人健康造成嚴重影響
# Change Classification Metrics
𝑃_𝑜𝑏𝑠𝑒𝑟𝑣𝑒𝑑: Probability of observed agreement (ie. the overall accuracy of the model)
𝑃_𝑏𝑦𝑐ℎ𝑎𝑛𝑐𝑒: Probability of agreement by chance (ie. the measure of the agreement between the model predictions and the actual class values as if happening by chance)
Kappa coefficient:
𝜅=(𝑃_𝑜𝑏𝑠𝑒𝑟𝑣𝑒𝑑−𝑃_𝑏𝑦𝑐ℎ𝑎𝑛𝑐𝑒)/(1−𝑃_𝑏𝑦𝑐ℎ𝑎𝑛𝑐𝑒 )
Ex:
TP:1 FP:1
FN:8 TN:90
Total=100
TP:1/100=0.01
TN:90/100=0.9
FN:8/100=0.08
FP:1/100=0.01
𝑃_𝑜𝑏𝑠𝑒𝑟𝑣𝑒𝑑= 0.01 + 0.9 = 0.91
𝑃_𝑏𝑦𝑐ℎ𝑎𝑛𝑐𝑒= 0.02(TP+FP) * 0.09(TP+FN) + 0.98(FN+TN) * 0.91(FP+TN) = 0.8936
𝜅=(𝑃_𝑜𝑏𝑠𝑒𝑟𝑣𝑒𝑑−𝑃_𝑏𝑦𝑐ℎ𝑎𝑛𝑐𝑒)/(1−𝑃_𝑏𝑦𝑐ℎ𝑎𝑛𝑐𝑒 )
= (0.91 −0.8936)/(1 −0.8936)
= 0.0164/0.1064=15.41%
Kappa系數(κ)用於衡量分類模型的預測結果和實際觀察結果之間的一致性,其值範圍通常在[−1,1] 之間。Kappa系數越接近1,表示模型預測結果和實際觀察結果的一致性越高,即模型效果越好
若資料集越 balanced, 𝜅 的值越大
# Resampling Dataset
**Over-sampling**
sampling with replacement :
把樣本從母體抽樣出來之後,再放回母體給下次的抽樣。意思就是同一筆資料可能出現多次。
SMOTE :
該算法通過選擇兩個或多個相似的樣本(使用距離度量)來生成合成樣本,並在不同屬性上通過隨機數量的擾動來產生新的樣本。如何選擇鄰居以及計算距離是改進這種算法的新方向。
1.選擇少數類別樣本:從少數類別中選擇一個樣本𝑥𝑖。
2.尋找鄰居:在少數類別中使用距離度量(如歐幾里得距離)找到該樣本的k個最近鄰居。
3.生成合成樣本:對於每個選定的鄰居樣本𝑥𝑖,𝑛𝑒𝑖𝑔ℎ𝑏𝑜𝑟,生成一個合成樣本。具體步驟如下:
* 對於樣本的每個屬性,計算兩個樣本間的差異。
* 將這個差異乘以一個介於0和1之間的隨機數,再加上原樣本的屬性值。
* 這樣,每個屬性就被隨機擾動了一個值,產生了一個新的合成樣本。
假設我們有一個少數類別樣本𝑥𝑖和它的鄰居𝑥𝑖,𝑛𝑒𝑖𝑔ℎ𝑏𝑜𝑟,它們在兩個特徵空間中的表示如下:
𝑥𝑖=(2,3)
𝑥𝑖,𝑛𝑒𝑖𝑔ℎ𝑏𝑜𝑟=(4,7)
為生成一個合成樣本,我們可以按以下步驟進行:
計算每個屬性的差異:
差異 =𝑥𝑖,𝑛𝑒𝑖𝑔ℎ𝑏𝑜𝑟−𝑥𝑖=(4−2,7−3)=(2,4)
對每個屬性乘以一個隨機數(假設為 0.5):
擾動值 =(2∗0.5,4∗0.5)=(1,2)
將擾動值加到原樣本上:
新的合成樣本 =𝑥𝑖+擾動值=(2+1,3+2)=(3,5)=x
因此,新的合成樣本為 (3,5)。
選擇鄰居和計算距離是改進SMOTE算法的關鍵方向
1.距離度量的選擇:
歐幾里得距離:最常用的距離度量,但對於高維數據和非線性數據效果較差。
曼哈頓距離:對於一些特定的數據分佈可能更有效。
馬氏距離:考慮了數據分佈的共變異數矩陣,對高維數據更穩定。
余弦相似度:適用於文本數據等高維稀疏數據。
2.鄰居選擇的策略:
隨機選擇:傳統SMOTE中隨機選擇k個鄰居,但可能包含噪聲數據。
聚類選擇:通過聚類方法(如KMeans)選擇同一聚類中的鄰居,減少噪聲影響。
加權選擇:根據樣本的重要性或相似性給予鄰居不同的權重。
**Under-sampling**
範例程式碼:
```
import random
import numpy as np
import pandas as pd
from imblearn.over_sampling import RandomOverSampler, SMOTE
from imblearn.under_sampling import RandomUnderSampler
from collections import Counter
import seaborn as sns
import matplotlib.pyplot as plt
# 設定隨機種子,使每次產生的資料相同
random.seed(2022)
np.random.seed(2022)
n1 = 200 # 第一類的數量(type1)
n2 = 20 # 第二類的數量(type2)
# 產生type1的data
x = np.random.normal(0, 0.5, n1)
y = np.random.normal(0, 1, n1)
data1 = pd.DataFrame({'label': ['type1'] * n1, 'x': x, 'y': y})
# 產生type2的data
x2 = np.random.normal(2, 0.5, n2)
y2 = np.random.normal(2, 1, n2)
data2 = pd.DataFrame({'label': ['type2'] * n2, 'x': x2, 'y': y2})
# 建立 imbalanced_data
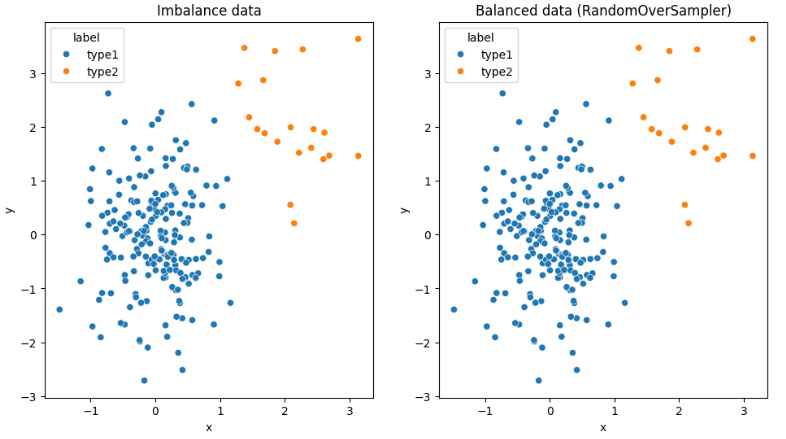
imbalance_data = pd.concat([data1, data2])
print(imbalance_data['label'].value_counts())
# type1 200
# type2 20
```
```
# 使用 RandomOverSampler 進行過採樣
oversample = RandomOverSampler(sampling_strategy='minority')
X_over, y_over = oversample.fit_resample(imbalance_data[['x', 'y']], imbalance_data['label'])
balanced_data_over = X_over
balanced_data_over['label'] = y_over
print(Counter(balanced_data_over['label']))
# Counter({'type1': 200, 'type2': 200})
```
```
# 使用 SMOTE 進行過採樣
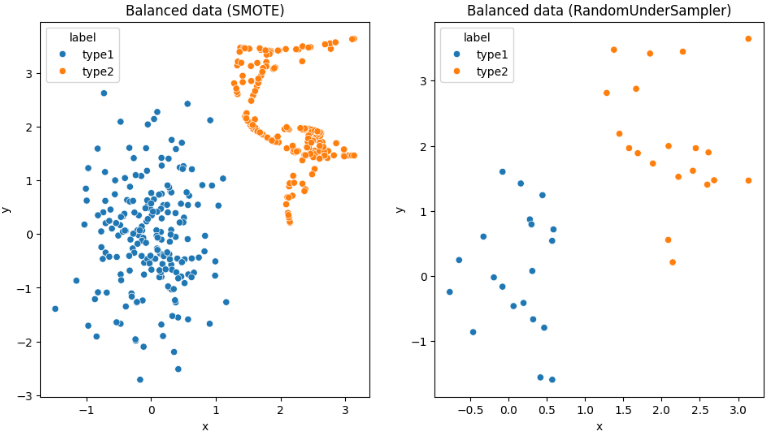
X_smote, label_smote = SMOTE(random_state=2022).fit_resample(imbalance_data[['x', 'y']], imbalance_data['label'])
balanced_data_smote = X_smote
balanced_data_smote['label'] = label_smote
print(Counter(balanced_data_smote['label']))
# Counter({'type1': 200, 'type2': 200})
```
```
# 使用 RandomUnderSampler 進行欠採樣
undersample = RandomUnderSampler(sampling_strategy='majority')
X_under, y_under = undersample.fit_resample(imbalance_data[['x', 'y']], imbalance_data['label'])
balanced_data_under = X_under
balanced_data_under['label'] = y_under
print(Counter(balanced_data_under['label']))
# Counter({'type1': 20, 'type2': 20})
```
```
# 繪圖
fig, axs = plt.subplots(ncols=4, figsize=(24, 6))
sns.scatterplot(data=imbalance_data, x='x', y='y', hue='label', ax=axs[0]).set(title='Imbalance data')
sns.scatterplot(data=balanced_data_over, x='x', y='y', hue='label', ax=axs[1]).set(title='Balanced data (RandomOverSampler)')
sns.scatterplot(data=balanced_data_smote, x='x', y='y', hue='label', ax=axs[2]).set(title='Balanced data (SMOTE)')
sns.scatterplot(data=balanced_data_under, x='x', y='y', hue='label', ax=axs[3]).set(title='Balanced data (RandomUnderSampler)')
plt.show()
```
 Sign in with Wallet
Sign in with Wallet
 Sign in with Wallet
Sign in with Wallet