---
tags: 偏鄉醫療進度報告
---
# 2022/07/14 偏鄉醫療進度報告
> 林興政、劉彥汝
## 心電圖(12導程)介紹
1. :bulb:**簡述**:
十二導程心電圖監視是由十個導極所組成的十二個導程,記錄**心臟於三維空間中不同角度的電氣活動,以了解心臟各區塊之狀況**。
2. [**心電圖檢查**](https://helloyishi.com.tw/heart-health/what-is-electrocardiography/):
> 心電圖是一種方便、快速的心臟檢測方法,且不需額外自費。
> 基本的心臟資訊如心跳速度、心臟電位變化等,都能由心電圖結果得知。
- 一般來說,出現以下症狀可能會需要接受心電圖檢查:
- 胸痛
- 呼吸困難
- 心悸
- 虛弱
- 頭暈、頭昏眼花
- 暈厥
3. **心電圖怎麼看 (PQRST波)**

- **<font color="#f00">P波</font>**:在1次完整的心臟電位變化中,出現的第一個偏離即稱為P波。這時心房會發生去極化造成心房收縮。
- **<font color="#f00">PR段</font>**:指的是P波到發生QRS波中間的時間段。
- **<font color="#f00">QRS波組</font>**:QRS並非固定的排序,在命名上,波組中第一個向下偏轉的波形稱為Q波,
若向上則稱為R波,而接續R波之後的負向偏轉稱為S波。值得注意的是,
QRS並非3者必然同時存在,也可能出現只有QR 、QS ,或甚至只有R波的可能性。
- **<font color="#f00">ST段</font>**:QRS波與T波中間的時間段。
- **<font color="#f00">T波</font>**:心室再極化造成心室舒張。
- **<font color="#f00">QT間隔</font>**:指的是心臟收縮到舒張結束的所需時間長度。有一種「長QT症候群」便是指QT間隔過長造成的心臟症狀。
4. 心電圖三大異常波型
- **Q波大幅下探**:代表曾有發生過心肌梗塞的狀況。
- **R波大幅上升**:可能代表有左心室肥大而引起波形放大。
- **T波倒置**:在正常的心電圖中,T波通常呈現正向偏轉,如果T發生負向偏轉,則可能代表心臟有缺血或曾發生或心肌梗塞的狀況。
5. **測量器具:**
- 10片電極貼片
- 心電圖儀器
6. **pros and cons:**
- pros:
- 非侵入式檢測且無輻射
- cons:
- 需要電極片等...專業儀器
## Heart Rate Variability (HRV) 心率變異率介紹 (或稱自律神經檢測)
1. :bulb:**簡述**:
一種**量測連續心跳速率變化程度**的方法,利用心跳速度的變化作為指標,
間接了解自律神經的活性狀態。
2. [**臨床應用**](http://www.westgarden.com.tw/medicenter_gallery/%E5%82%BE%E8%81%BD%E9%AB%94%E5%85%A7%E6%B7%B1%E5%B1%A4%E7%9A%84%E8%81%B2%E9%9F%B3-%E8%87%AA%E5%BE%8B%E7%A5%9E%E7%B6%93%E6%AA%A2%E6%B8%AC%E5%84%80%E4%B9%8B%E8%87%A8%E5%BA%8A%E6%87%89%E7%94%A8%E8%A5%BF/):
> 當心率變異數值愈差,未來罹患或死於心血管疾病的機率愈高。
- 當心率變異不正常,代表自律神經功能不好,也反映可能隱藏著的身心不健康。
- 預測發生心血管疾病病人的長期存活率
- 可做為個人整體健康情形的重要指標
:accept: 適用對象為不明原因失眠、心悸、胸悶、倦怠、頭暈、腸胃不適等族群
3. **測量器具:**
- 一台可以測量脈搏的儀器

- UNISAGE HRV檢測儀(台灣本土的寰碩公司所研發製造)
4. **pros and cons:**
- pros:
- 檢測過程約5~10分鐘
- 非侵入式檢測且無輻射
- cons:
- 某些特定狀況不適合接受HVR檢測,包括:嬰兒、本身裝有心律調節器者、已知有心律不整之病症、心臟已裝有心導管支架者、長期服用鎮定劑或是興奮劑且無法停藥者。
## 心律變異度(HRV) 名詞介紹
> [HRV wiki](https://zh.m.wikipedia.org/zhtw/%E5%BF%83%E7%8E%87%E8%AE%8A%E7%95%B0%E5%88%86%E6%9E%90)
### <font color="#f00">time domain</font>
> R-R代表 R心跳 和 R+1心跳 間隔的時間
- **bpm** -> the amount of heart <font color="#f00">b</font>eats <font color="#f00">p</font>er <font color="#f00">m</font>inute, 每分鐘的心跳次數
- **ibi** -> <font color="#f00">i</font>nter-<font color="#f00">b</font>eat <font color="#f00">i</font>nterval, 心跳間隔的平均距離
- **sdnn** -> 心跳間隔的標準差
> 
- **sdsd** -> 相鄰 R-R 區間之間連續差異的標準差
> 
- **rmssd** -> 相鄰 R-R 區間之間連續差的均方根
> 
- **nn20** -> 心電圖中所有每對相鄰正常心跳時間間隔,差距超過20ms的數目
> nn -> normal to normal
> 也就是我們所說的RR interval(心電圖QRS的R,兩個R相間隔的時間算一次心跳時間)
- **nn50** -> 心電圖中所有每對相鄰正常心跳時間間隔,差距超過50ms的數目
- **pnn20** -> NN20數目除以量測之心電圖中所有的正常心跳間隔總數
- **pnn50** -> NN50數目除以量測之心電圖中所有的正常心跳間隔總數
- **median** absolute deviation, MAD (中位數絕對離差)
> 原數據減去中位數後,得到的新數據的絕對值的中位數
- [Poincare analysis](https://rightweightleeding.wordpress.com/2017/09/27/hrv%E9%BE%90%E5%8A%A0%E8%90%8A%E5%9C%96-poincare-plot%E5%88%86%E6%9E%90/) (SD1, SD2, S, SD1/SD2) 龐加萊分析
- [Poincare plotting](https://en.wikipedia.org/wiki/Poincar%C3%A9_plot) 龐加萊圖
### <font color="#f00">frequency domain</font>
- very low frequency component (0.0033–0.04 Hz), VLF
- low frequency component (0.04–0.15 Hz), LF
- high frequency component (0.15–0.4 Hz), HF
- lf/hf ratio, LF/HF
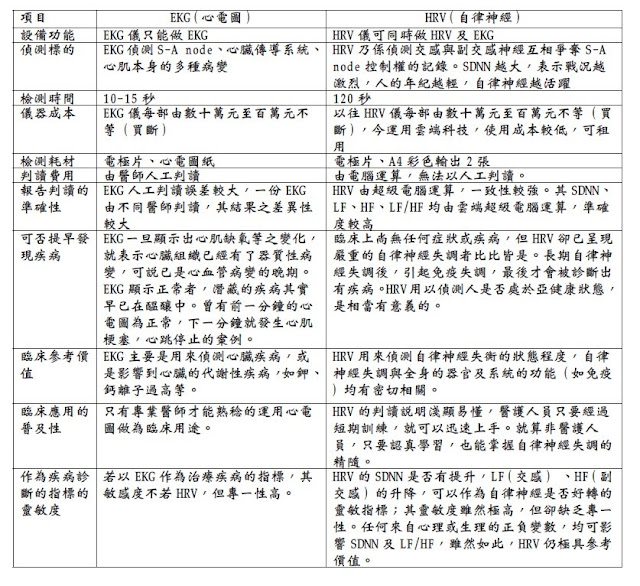
## 自律神經(HRV)檢測 vs 心電圖(EKG)
> [link](http://hrvtw.blogspot.com/2010/09/hrv_13.html)
- 自律神經(HRV)檢測的方法與做心電圖(EKG)原理步驟方法完全一致,做出來的同樣都是心電圖,只是<font color="#f00">解讀此心電圖的角度、分析重點</font>不同而已。
- EKG是根據心電圖的形態(Morphplogy)、結構來判定
ex:竇房結(S-A node)、房室結(A-V node)、房室束、普金氏纖維有無阻滯異常;心肌有無病變、壞死、肥大以及心軸有無偏移
- HRV則是對同樣一份心電圖(EKG),另外一種角度的判讀,經由觀測自律神經對竇房結(S-A node)調控的程度來判定人的自律神經生理活性(時域:SDNN)及交感副交感強度(頻域:LF、HF)
- 比較表:
> 
## heartpy套件介紹
### 數據
- data.csv
> 
- data2.csv
> 
>
1. 對於數據來源,套件開發者並未說明
2. data.csv的數據過於理想,可以推測為開發者在最理想狀況下的思路與分析
3. data2.csv的數據為開發者預測可能發生情況(儀器移動、測試者移動)去做推測與分析
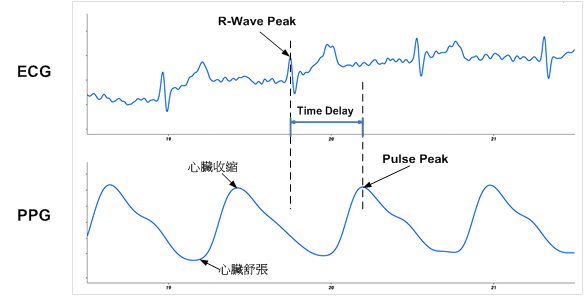
### ECG/PPG數據分析方法
- **心電圖(ECG) vs 光體積變化描記圖法(PPG)**:
> 
:::info
:bulb:**Hint**
從上圖可以發現,雖然標記點並不在同一個時間點上。
不過對於計算週期性的數據並無影響,兩者在一次心臟跳動中恰好都有一個最高點可以紀錄,因此本套件可以分析兩種數據。
:::
### heartpy 實作
1. **part1 - 利用 heartpy 套件偵測範例資料的高峰與計算 BPM**
- 先以理想的狀態去思考(data.csv)
```python
def rolmean(dataset, hrw, fs):
mov_avg = dataset['hart'].rolling(int(hrw*fs)).mean()
avg_hr = (np.mean(dataset.hart))
mov_avg = [avg_hr if math.isnan(x) else x for x in mov_avg]
mov_avg = [x*1.2 for x in mov_avg]
dataset['hart_rollingmean'] = mov_avg
```
```python
def detect_peaks(dataset):
window = []
peaklist = []
listpos = 0
for datapoint in dataset.hart:
rollingmean = dataset.hart_rollingmean[listpos]
if (datapoint < rollingmean) and (len(window) < 1):
listpos += 1
elif (datapoint > rollingmean):
window.append(datapoint)
listpos += 1
else:
beatposition = listpos - len(window) + (window.index(max(window)))
peaklist.append(beatposition)
window = []
listpos += 1
measures['peaklist'] = peaklist
measures['ybeat'] = [dataset.hart[x] for x in peaklist]
```
1. 先畫一條移動平均線
2. 標記心律信號位於移動平均線上方的ROI
3. 在每個ROI中找到最高點
> 
2. **part2 - 從心率信號中提取複雜的測量值**
- 計算測量值:
>
3. **part3 - 新增 filters 過濾雜訊,利用 dynamoc threshold 增強檢測能力**
- 再思考其他狀況(測量中儀器改變測量位置、患者打噴嚏、移動或說話等干擾因素):
detect_peaks()函數將在心率信號從小於移動平均線變為變為等於而連續至少兩個數據點都沒有移動到其上方時中斷,發生這種情況的最可能情況是信號在一段時間內下降到零。然後,該函數將跳到else語句,並嘗試檢測ROI中的峰值,但是由於信號從未上升到移動平均線之上,因此未標記ROI,因此max(window)拋出錯誤,因為空列表沒有最大值。
```python
def detect_peaks(dataset):
window = []
peaklist = []
listpos = 0
for datapoint in dataset.hart:
rollingmean = dataset.hart_rollingmean[listpos]
if (datapoint <= rollingmean) and (len(window) <= 1):
listpos += 1
elif (datapoint > rollingmean):
window.append(datapoint)
listpos += 1
else:
beatposition = listpos - len(window) + (window.index(max(window)))
peaklist.append(beatposition)
window = []
listpos += 1
measures['peaklist'] = peaklist
measures['ybeat'] = [dataset.hart[x] for x in peaklist]
```
>
- Sampling Frequency - 讓所有資料都變成同樣的頻率
- Signal Filtering - 去除雜訊
```python
def filter_signal(data, cutoff, sample_rate, order=2, filtertype='lowpass', return_top = False):
if filtertype.lower() == 'lowpass':
b, a = butter_lowpass(cutoff, sample_rate, order=order)
elif filtertype.lower() == 'highpass':
b, a = butter_highpass(cutoff, sample_rate, order=order)
elif filtertype.lower() == 'bandpass':
assert type(cutoff) == tuple or list or np.array,
b, a = butter_bandpass(cutoff[0], cutoff[1], sample_rate, order=order)
elif filtertype.lower() == 'notch':
b, a = iirnotch(cutoff, Q = 0.005, fs = sample_rate)
else:
raise ValueError('filtertype: %s is unknown, available are: lowpass, highpass, bandpass, and notch' %filtertype)
filtered_data = filtfilt(b, a, data)
if return_top:
return np.clip(filtered_data, a_min = 0, a_max = None)
else:
return filtered_data
```
- Improving Detection Accuracy With a Dynamic Threshold
- Finding Incorrectly Detected Peaks
4. part4 - Still processing
 Sign in with Wallet
Sign in with Wallet
 Sign in with Wallet
Sign in with Wallet